RNA分子通过错综复杂的碱基配对模式折叠成二级结构,这些二级结构是RNA行使催化、配体结合和支架等功能的基础。准确地测定RNA的二级结构对于理解其生物学功能以及调控机制至关重要。近年来,化学小分子探针被广泛应用于RNA二级结构的探测,其中,selective 2´-hydroxyl acylation analyzed by primer extension(SHAPE)技术利用亲电试剂对不同状态碱基(配对或未配对)的修饰活性的差异来实现对RNA二级结构的探测。在SHAPE数据的分析中,如何合理地将SHAPE数据整合进结构预测算法是一个关键的问题。常用的工具,如RNAstructure,通常将SHAPE值转化成每个配对核苷酸各自的伪自由能约束,进而整合进热力学预测模型中预测出最终的结构。这类整合方法虽然显著地提升了RNA二级结构预测的准确度,但提升程度仍不能满足研究者的需求,尤其是对长RNA的结构预测和对结构动态变化的捕捉。
清华大学生命科学学院薛毅课题组致力于开发新的计算及实验方法来研究生物大分子的结构与动态。在本项研究中,他们通过统计分析发现,对于同一类型(发卡环、内环和凸环)和长度的环结构元件而言,其通常具有某种特征性的SHAPE模式,即在某些核苷酸位置之间存在保守的SHAPE值高低趋势。通过对环元件的三级结构分析,他们发现上述特征性SHAPE模式可以反映出核苷酸对糖环构象的偏好性。为了利用这种特征性SHAPE模式来提升RNA二级结构预测的准确度,他们提出了SHAPELoop方法。该方法通过“评估-替换”的策略对传统能量模型预测出的结构(称为“指导结构”)进行改进,以达到SHAPELoop预测出的结构比指导结构更加符合特征性SHAPE模式的目的(图1)。
利用上述策略,SHAPELoop在预测的准确度上超过了传统的伪自由能预测模型(RNAstructure和RME)以及“采样-选择”模型(SeqFold)。此外,他们也发现特征性SHAPE模式可以帮助鉴定假结结构以及RNA二级结构的动态变化。
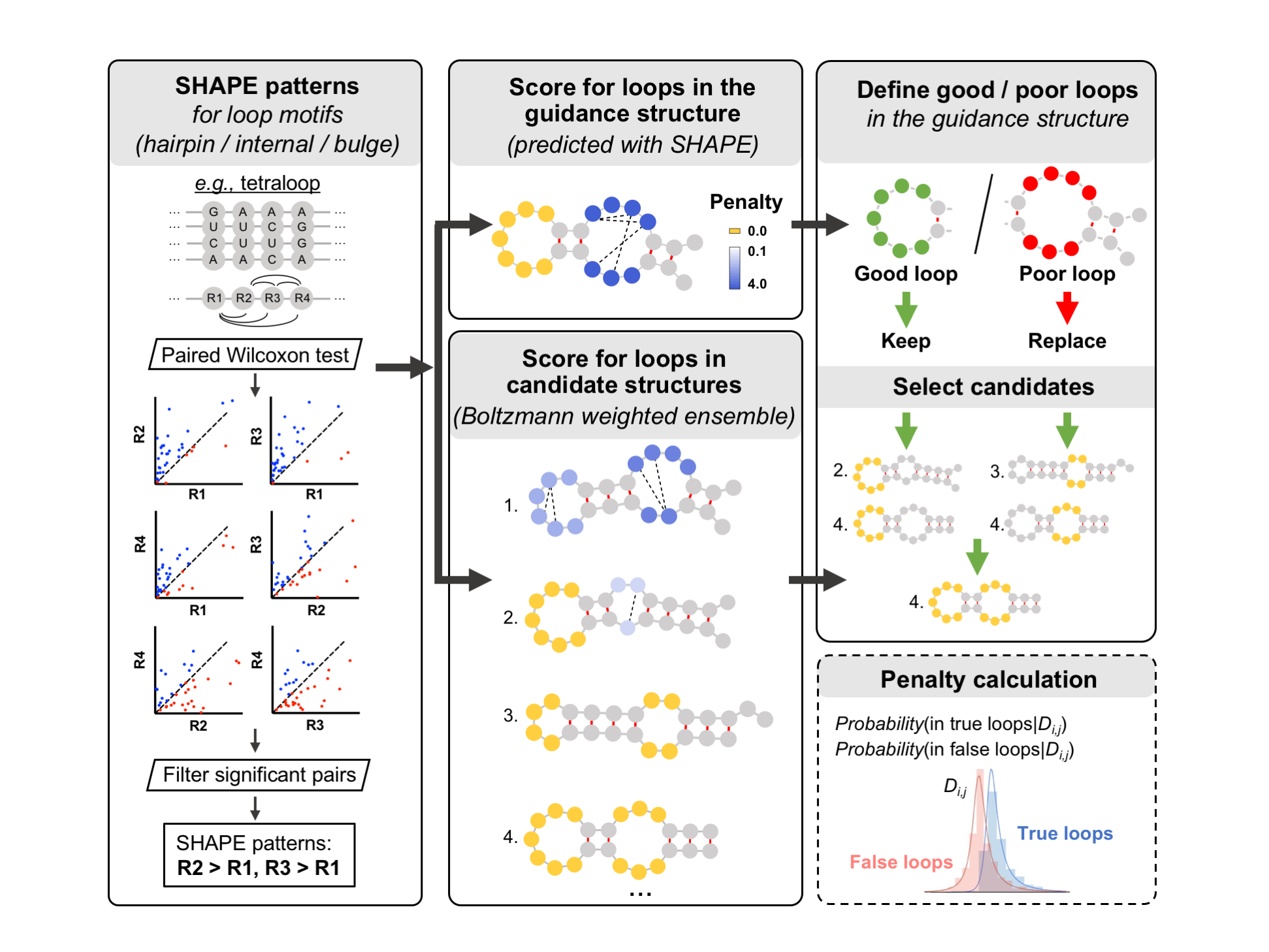
图1. SHAPELoop方法框架
该研究工作于2021年4月13日在《核酸研究》(Nucleic Acids Research)杂志上发表,题为“环结构元件的特征性化学探测数据模式及其对RNA二级结构预测准确度的改善”(Characteristic chemical probing patterns of loop motifs improve prediction accuracy of RNA secondary structures),原文链接为:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab250/6225227。清华大学生命学院薛毅研究员为本文通讯作者,清华大学生命学院博士生曹静怡为本文第一作者。本研究得到了清华-北大生命科学联合中心和北京市结构生物学高精尖创新中心的经费支持。