新型冠状病毒是全球新冠肺炎大爆发的罪魁祸首(Perlman, 2020)。迄今为止,已经感染了1亿多人,死亡人数超过250万,给人类社会造成了巨大灾难。新冠病毒是单链RNA病毒,其RNA基因组是调控病毒感染的重要因子。新冠病毒的RNA基因组非常小,所编码的蛋白质数目也很有限,病毒的生命周期极大依赖宿主细胞中的蛋白质因子来完成(Gordon et al., 2020; Schmidt et al., 2020)。解析新冠病毒RNA基因组的在宿主细胞内的分子结构,确定与其相互作用的宿主因子,有助于开发治疗新冠肺炎的特效药物。
近日,清华大学生命科学学院张强锋教授课题组与中国医学科学院王健伟教授课题组、清华大学医学院丁强教授课题组合作,解析了人细胞内新冠病毒RNA的全基因组结构。基于细胞内的病毒RNA结构数据,张强锋课题组依靠新开发的人工智能算法工具PrismNet,预测了大量结合新冠RNA的宿主蛋白质因子。令人振奋的是,研究团队发现,一些宿主因子已有的靶向药物,可以经过重定位,在细胞内抑制新冠病毒感染,是抗新冠肺炎的潜在候选药物(图1)。
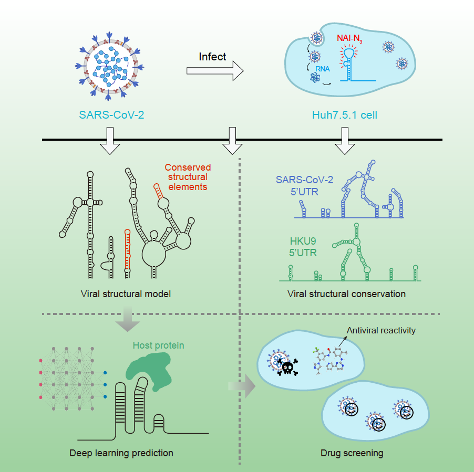
图1 解析新冠病毒体内RNA结构,并基于结构发现相关宿主因子,筛选潜在抗病毒重定位药物(图源自Sun et. al., 2021a)
RNA结构是RNA功能和调控的基础。长期以来,科研工作者通过X射线晶体衍射、核磁共振、冷冻电子显微镜等方法解析了大量RNA结构,揭示了各种RNA结构与功能关系。近年来一些新的结构解析方法给开发出来,结合细胞内的人为RNA化学修饰和深度测序技术,在全转录组水平检测细胞内的RNA二级结构(Spitale et al., 2015)。这些新的RNA高通量技术,揭示了大量RNA结构参与转录后调控的规律和机制。
张强锋课题组自建立起一直致力于研究RNA结构,专注于新技术的开发和应用,并进一步利用这些新方法深入理解RNA结构的科学意义及医学应用。在之前的工作中,张强锋课题组利用两种RNA结构新技术(icSHAPE技术与PARIS技术)解析了寨卡病毒的RNA基因组结构。寨卡病毒在2007年爆发,其病毒基因组可以大致分为流行的亚洲株系和非流行的非洲株系。该课题组的研究发现了一个在亚洲株系中特异存在的RNA结构元件,实验验证表明该结构元件可能会提高病毒的感染力,为流行病毒株系导致的病毒大流行提供了新的解释(Li et al., 2018)。
张强锋课题组还致力于开发新的人工智能方法来解决复杂的生物学问题。在一个与该新冠病毒RNA结构研究同时发表的工作中,该课题组通过整合对应的细胞内RNA结构信息和RBP结合信息,开发了一个可以准确预测RNA结合蛋白(RBP)的结合位点及其变化的深度神经网络PrismNet(图2)。RBP在转录、RNA代谢以及翻译过程中起重要的调控作用。CLIP实验技术作为RNA研究最重要的技术之一,可以解析RBP在整个转录组上的结合图谱,是系统理解一个RBP功能及其调控机制的基础。多年的科学研究积累了大约200个基于CLIP实验的RBP转录组结合图谱,是研究RNA调控的重要资源。PrismNet的一个重要应用价值在于大大扩充了这个资源。比如,对于任意一个RBP,只要在任意一个细胞背景下做了CLIP实验,PrismNet就可以通过构建准确的深度神经网络模型,把该RBP的结合信息外推到其他解析了RNA结构的细胞背景中(Sun et al., 2021b)。
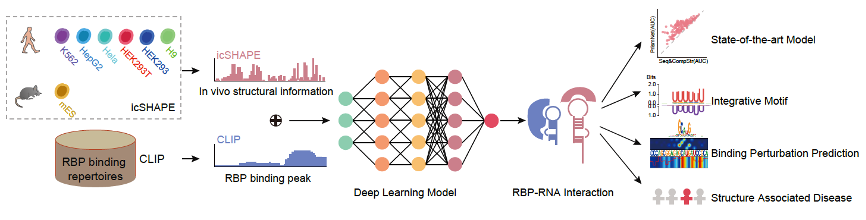
图2 人工智能方法PrismNet的构建及应用(图源自Sun et. al., 2021b)
疫情爆发后,张强锋课题组迅速行动,与中国医学科学院王健伟课题组以及清华大学丁强课题组通力合作,致力于解析新冠病毒感染的分子机制,发现抗病毒的特效药物。基于课题组之前的技术优势,张强锋课题组利用icSHAPE技术解析了感染细胞内新冠病毒RNA基因组的二级结构图谱并搭建了二级结构模型。通过结构模型,作者找到了大量保守的RNA结构元件,并通过反义多聚核酸(ASO)干扰、结构突变等实验手段验证了一些RNA元件对病毒生长的作用。
更重要的是,课题组利用解析得到的新冠病毒的RNA结构和上文提到的人工智能方法PrismNet,准确预测了多个与新冠病毒RNA相互作用的宿主蛋白,并对其中一部分相互作用及其功能相关性进行了实验验证。针对这些宿主因子,合作团队找到了多个的市场上已有的靶向药物。实验表明,经过这些药物处理的细胞感染新冠病毒的能力大大减弱了。这意味着,这些重定位药物可以为治疗新冠肺炎提供新的潜在候选治疗方案(Sun et al., 2021a)。
该研究工作在Cell杂志和Cell Research杂志上同时发表。其中新冠病毒RNA基因组结构解析和药物重定向的工作发表在Cell杂志上(2月9日),题为“通过细胞内新冠病毒RNA结构发现靶向宿主蛋白的重定位药物”(In vivo structural characterization of the SARS-CoV-2 RNA genome identifies host proteins vulnerable to repurposed drugs),原文链接为:https://www.cell.com/cell/fulltext/S0092-8674(21)00158-6;研究中所使用的预测细胞内RNA-蛋白质相互作用的人工智能方法发表在Cell Research杂志上(2月23日),题为“使用人工智能方法基于细胞内RNA结构预测蛋白质-RNA动态相互作用”(Predicting dynamic cellular protein–RNA interactions by deep learning using in vivo RNA structures),原文链接为:https://doi.org/10.1038/s41422-021-00476-y。
清华大学张强锋研究员、中国医学科学院王健伟教授以及清华大学丁强研究员为新冠病毒论文的共同通讯作者;清华大学博士后孙磊、清华大学博士生李盼、清华大学博士生鞠晓辉、中国医学科学院博士后饶健、清华大学博士生黄文泽为该论文共同第一作者。同时,张强锋研究员还是人工智能方法论文的通讯作者;清华大学博士后孙磊、博士生徐魁、黄文泽等为该论文共同第一作者。研究工作获得国家自然科学基金委、科技部重点研发计划、清华大学春风基金,北京结构生物学高精尖创新中心、清华-北大生命联合中心等资助。
参考文献:
Gordon, D.E., Jang, G.M., Bouhaddou, M., Xu, J., Obernier, K., White, K.M., O'Meara, M.J., Rezelj, V.V., Guo, J.Z., Swaney, D.L., et al. (2020). A SARS-CoV-2 protein interaction map reveals targets for drug repurposing. Nature 583, 459-468.
Li, P., Wei, Y., Mei, M., Tang, L., Sun, L., Huang, W., Zhou, J., Zou, C., Zhang, S., Qin, C.F., et al. (2018). Integrative Analysis of Zika Virus Genome RNA Structure Reveals Critical Determinants of Viral Infectivity. Cell Host Microbe 24, 875-886 e875.
Perlman, S. (2020). Another Decade, Another Coronavirus. N Engl J Med 382, 760-762.
Schmidt, N., Lareau, C.A., Keshishian, H., Ganskih, S., Schneider, C., Hennig, T., Melanson, R., Werner, S., Wei, Y., Zimmer, M., et al. (2020). The SARS-CoV-2 RNA-protein interactome in infected human cells. Nat Microbiol.
Spitale, R.C., Flynn, R.A., Zhang, Q.C., Crisalli, P., Lee, B., Jung, J.W., Kuchelmeister, H.Y., Batista, P.J., Torre, E.A., Kool, E.T., et al. (2015). Structural imprints in vivo decode RNA regulatory mechanisms. Nature 519, 486-490.
Sun, L., Li, P., Ju, X., Rao, J., Huang, W., Zhang, S., Xiong, T., Xu, K., Zhou, X., Ren, L., et al. (2021a). In vivo structural characterization of the whole SARS-CoV-2 RNA genome identifies host cell target proteins vulnerable to re-purposed drugs. Cell https://doi.org/10.1016/j.cell.2021.02.008.
Sun, L., Xu, K., Huang, W., Yang, Y.T., Li, P., Tang, L., Xiong, T., and Zhang, Q.C. (2021b). Predicting dynamic cellular protein-RNA interactions using deep learning and in vivo RNA structure. Cell Research, https://doi.org/10.1038/s41422-41021-00476-y.