2020年1月13日,清华大学生命科学学院李雪明研究组在《自然通讯》(Nature Communications)杂志在线发表题为“curli菌毛生成系统的组装和底物识别”(Assembly and substrate recognition of curli biogenesis system)的研究论文。该论文报道了细菌curli菌毛生成系统(也称细菌八型分泌系统)膜上通道复合物的近原子分辨率结构,结合结构生物学手段和生化研究揭示了其新的组装机制和底物识别机制。同时,该研究开发了一个新的多肽抑制剂,为之后curli生成系统抑制剂的研发提供了新思路。
为了在宿主体内或者环境中更好的定植和生存,许多细菌选择聚集在一起形成由蛋白,核酸以及多糖形成的亲水基质包裹的封闭群落。这些由大量亲水大分子包裹的基质叫作生物被膜。细菌的生物被膜在很多感染性疾病中充当重要的致病因素,例如屏蔽型静脉导管,人工瓣膜感染性心内膜炎,以及Foley导管相关性尿路感染。curli菌毛作为生物被膜的重要组成部分,研究curli菌毛生成系统的组装和分泌机制对发展生物被膜相关疾病的治疗方法和治疗药物至关重要。curli生成系统由7个curli特异性相关基因(CsgA,CsgB,CsgC,CsgD,CsgE,CsgF和CsgG)组成,CsgA和CsgB是八型分泌系统的底物,CsgE,CsgF以及CsgG是分泌通道复合物的组成成分,负责特异性识别和分泌天然底物CsgA和CsgB。目前,对于通道复合物是如何组装以及底物CsgA是如何被通道复合物识别并分泌的机制并不清楚。
李雪明课题组通过冷冻电镜单颗粒重构的方法解析了大肠杆菌来源的curli生成系统膜上通道复合物CsgFG以及结合了底物的通道符合物CsgFG-CsgAN22的高分辨率结构,分辨率分别达到3.38 A 和3.34 A。CsgFG复合物的结构显示CsgF与CsgG形成9:9结合的双孔道复合物,CsgF的N端(CsgFN)插入到CsgG的b桶内部与CsgG相互作用。体外蛋白结合实验验证CsgFN与CsgG相互作用,CsgF的C端(CsgFC)与CsgB相互作用。进一步的体外蛋白结合实验显示CsgB结合CsgA,CsgF不结合CsgA。综合电镜结构和体外蛋白结合实验结果,我们提出了CsgG-CsgFN-CsgFC-CsgB-CsgA的组装方式,阐明了curli纤维如何结合到细菌表面的机制。此外,通过将CsgF的孔道氨基酸Asn17突变成不同侧链性质的氨基酸,我们发现疏水和带电氨基酸不利于亲水底物的转运,而亲水不带电的氨基酸更有利于底物转运。当把Asn17突变成长链的Arg,缩小了孔道的大小,同样也会影响底物的分泌。基于该实验结果,我们设计了一个多肽抑制剂PeptideNN17R,刚果红实验结果显示该抑制剂具有较强的抑制作用。
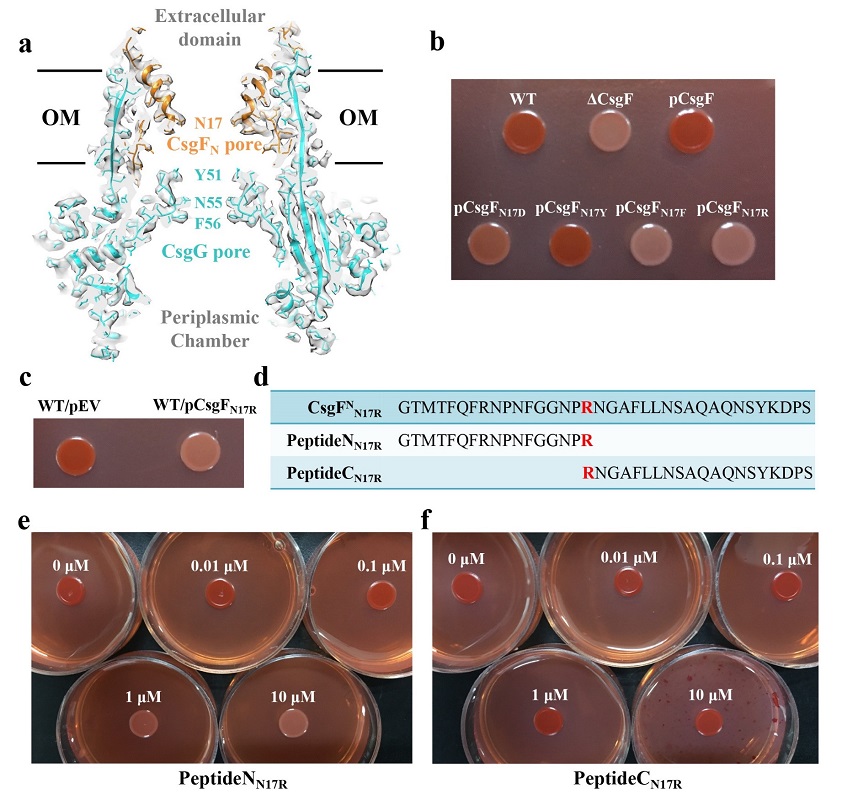
CsgFG的双孔道结构和多肽抑制剂
我们解析的CsgFG-CsgAN22的结构中显示CsgA的N末端6个氨基酸能与CsgG通道的外表面结合。多序列比对和单突变的刚果红实验结果显示保守的Val3和Gln5对结合CsgG和分泌底物CsgA至关重要。体外蛋白结合实验结果进一步发现CsgE能结合CsgA的R1-5结构域。CsgE的敲除菌株失去刚果红的表型,而过表达CsgG蛋白后刚果红表型得到恢复,这说明CsgE对底物分泌不是必须的,而是能提到转运效率,综合以上实验结果,我们提出了CsgA的识别转运模型。
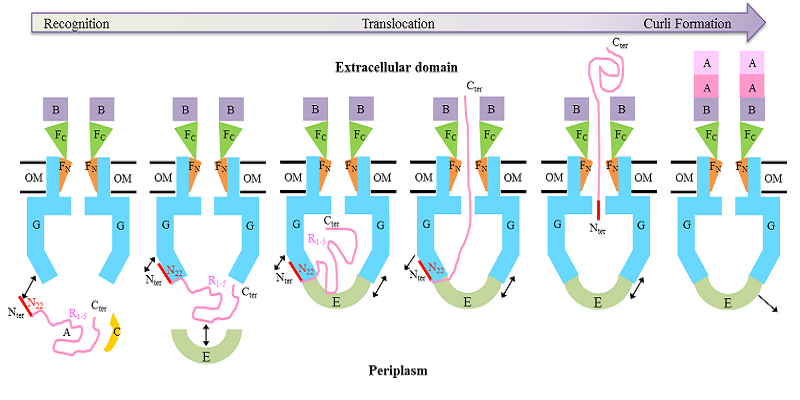
CsgA的识别转运模型
清华大学生命科学学院李雪明副教授为本文的通讯作者。清华大学生命学院2019届博士毕业生严照峰,清华大学PTN项目2019届博士毕业生尹孟和清华大学生命学院2018级博士生陈佳楠为本文的共同第一作者。该工作的冷冻电镜数据采集在国家蛋白质科学设施(北京)的清华大学冷冻电镜平台完成,数据处理在国家蛋白质科学设施(北京)清华大学生物计算平台完成。该工作获得了生命科学联合中心,北京市结构生物学高精尖创新中心,国家自然科学基金委面上项目和科技部重点研发计划的经费支持。
论文链接:https://www.nature.com/articles/s41467-019-14145-7