2020年6月17日,清华大学生命科学学院王新泉教授研究组在《自然通讯》(Nature Communications)在线发表题为《HKU2和SADS-CoV刺突蛋白的冷冻电镜结构为冠状病毒的进化提供了线索》(Cryo-EM structures of HKU2 and SADS-CoV spike glycoproteins provide insights into coronavirus evolution)的研究论文。猪冠状病毒SADS-CoV于2017年在中国广东的几家猪场被发现,其基因组与蝙蝠冠状病毒HKU2的同源性约为95%。本文报道了HKU2和SADS-CoV刺突蛋白三聚体2.38 A和2.83 A辨率的冷冻电镜结构(图一),并系统地比较了HKU2/SADS-CoV与其他α、β、γ和δ冠状病毒刺突蛋白中的各个结构域,表明HKU2/SADS-CoV的NTD和CTD分别代表了冠状病毒刺突蛋白的原始特征。这些结果在结构上揭示了HKU2/SADS-CoV(属于α冠状病毒属)与β冠状病毒属之间的密切进化关系,为冠状病毒的进化和跨物种传播提供了重要的线索。
冠状病毒包括α、β、γ和δ四个属,在人类、其他哺乳动物和鸟类中的感染可引起不同程度的呼吸、消化和神经系统疾病。人SARS、MERS和新型冠状病毒的传播对全球健康构成了严重威胁。冠状病毒感染也是家畜养殖业的重要威胁,以猪流行性腹泻病毒(PEDV)为代表的猪冠状病毒每周夺去10万只仔猪的生命,同时新型猪冠状病毒也在不断被发现,例如2017年分离的猪急性腹泻综合征冠状病毒(SADS-CoV,又称SeACoV和PEAV)在中国广东省的几家养猪场引起了仔猪严重水样腹泻,死亡率高达90%,造成了大量的经济损失。SADS-CoV是一种α属冠状病毒,在α属中具有代表性的成员有猪流行性腹泻病毒(PEDV)、猪传染性胃肠炎冠状病毒(TGEV)、猪呼吸道冠状病毒(PRCV)、猫传染性腹膜炎病毒(FIPV)、人NL63和229E冠状病毒(HCoV-NL63和HCoV-229E);β属的代表性成员包括小鼠肝炎冠状病毒(MHV)、牛冠状病毒(BCoV)、SARS-CoV、MERS-CoV、HCoV-OC43、HCoV-HKU1和新型冠状病毒;γ属的代表是禽传染性支气管炎病毒(IBV);δ属的代表是猪源deltacoronavirus(PdCoV)。
通过基因重组和/或突变促进的跨物种传播是冠状病毒宿主范围扩大的基础。蝙蝠是多种不同的α-和β-冠状病毒的自然宿主,它们通过重组和/或突变带来巨大的种间传播潜力。有关基因进化、受体结合和发病机制的数据表明,人类SARS-CoV-2、SARS-CoV和MERS-CoV最有可能来源于蝙蝠。新鉴定的猪SADS-CoV与蝙蝠冠状病毒HKU2的序列同源性约为95%,这也进一步强调了冠状病毒跨种传播的严重后果。
SADS-CoV刺突蛋白(1130个氨基酸残基)和HKU2刺突蛋白(1128个氨基酸残基)是最短的冠状病毒刺突蛋白之一,它们与其他已知冠状病毒刺突蛋白的氨基酸序列同源性低于28%,表明HKU2和SADS-CoV在进化中的特殊性。通过原子分辨率水平的冷冻电镜结构测定和比较,王新泉实验室发现α-冠状病毒刺突蛋白NTD有两种亚型,HKU2/SADS-CoV的NTD属于I亚型,而HCoV-229E的NTD属于II亚型,这两个亚型代表了NTD的原始结构,可能是其他属冠状病毒NTD的进化祖先。HKU2/SADS-CoV的CTD是一个包含5个反向排列strand的单层β-sheet,β-冠状病毒的CTD也具有类似的单层核心结构,目前已知的β-冠状病毒CTD的受体结合序列都在一个插入核心结构中的受体结合区(receptor-binding motif,RBM)内,但RBM在HKU2/SADS-CoV的 CTD中被一段短loop取代(图二)。该结果说明HKU2/SADS-CoV的CTD代表单层CTD的原始结构,而β-冠状病毒中RBM的插入可能是进化过程中重组事件的结果。值得注意的是,来自其他α-冠状病毒、γ-冠状病毒IBV和δ-冠状病毒PdCoV的CTD均由两层β-sheet组成的双层亚型(图二)。这些结果进一步证明HKU2可能是α-冠状病毒基因组骨架与未鉴别的原始β-冠状病毒的刺突蛋白基因进行重组的结果。
通过比较HKU2和SADS-CoV与其他冠状病毒刺突蛋白的结构,王新泉实验室发现在进化过程中,多个NTD可以重组进入同一个冠状病毒中(主要表现在α-冠状病毒),额外的loop区可以重组进入NTD的核心结构,受体结合区(RBM)可以重组进入CTD的核心结构。这些现象表明,结构域在进化过程中可以逐渐招募新的亚结构域,以进行跨物种传播并适应不同的宿主免疫系统,为病毒与宿主的协同进化提供了一个生动的例子。
清华大学生命科学学院王新泉教授研究组的博士生余锦芳为本文第一作者,谯淑元和郭润域为共同作者,王新泉教授为通讯作者。电镜数据采集和计算工作在国家蛋白质科学研究(北京)设施的冷冻电镜平台和生物计算平台完成。本工作获得北京市结构生物学高精尖创新中心、北京市生物结构前沿研究中心、清华大学自主科研基金、蛋白质科学教育部重点实验室、科技部重大科学研究计划专项和国家自然科学基金等的大力支持。
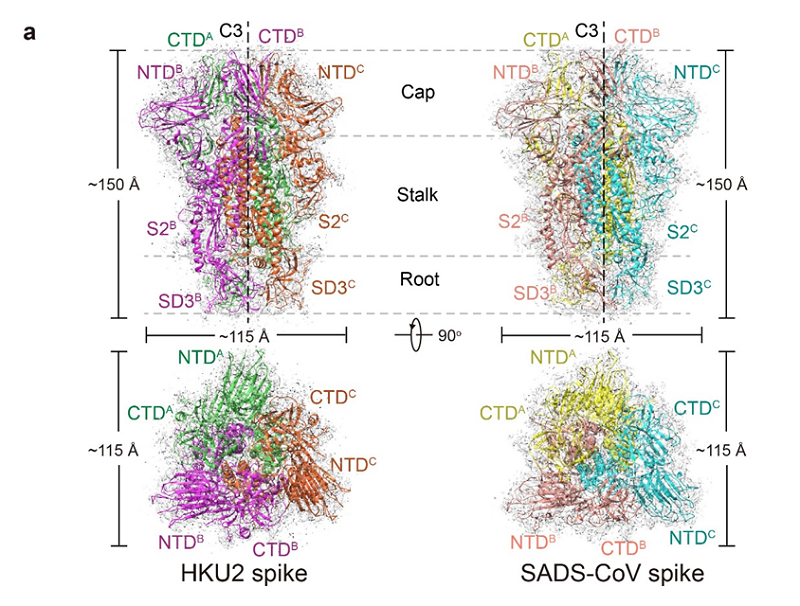
图一、HKU2和SADS-CoV的刺突蛋白三维结构
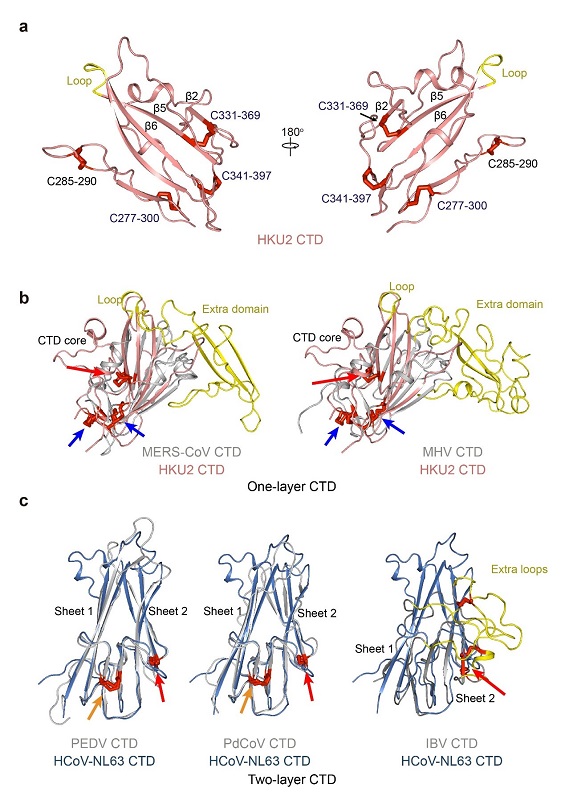
图二、HKU2与其他种属冠状病毒CTD的比较
论文链接:https://www.nature.com/articles/s41467-020-16876-4#Ack1