螺旋组装体(helical structure)广泛存在于细胞环境中,在细胞形态维持、物质运输、信息传递等的多种生物过程中扮演着重要角色,其三维结构的解析对于揭示相关的生物过程机制有重要的意义。螺旋参数的测定是实现螺旋结构三维结构解析的关键基础。目前常用的测定方法是层线法,通过对螺旋组装体电镜图像的功率谱中的线段状衍射图样进行测量与标定,以实现螺旋参数的解算。在实际应用中,冷冻电镜(cryo-electron microscopy,Cryo-EM)图像的极低信噪比(signal-to-noise rate, SNR)、螺旋组装体的柔性形变以及功率谱中的贝塞尔重叠(Bessel overlap)现象等问题,容易导致层线的模糊与偏移,使得该类方法的应用具有极大挑战性。此外,传统的层线法主要针对单颗粒图像设计,不适用于冷冻电子断层图像,制约了在原位结构解析中的应用。
2025年7月17日,清华大学生命科学学院李雪明副教授课题组和清华大学电子工程系沈渊教授课题组联合在《结构》(Structure)杂志在线发表研究论文,题目为“基于柱面展开的螺旋参数精确测量方法”(Accurate helical parameter estimation based on cylindrical unrolling)。该论文报道了一种名为HELIS(HELix Is Simple)的方法以及同名的软件,依托柱面晶格分析实现冷冻电子断层图像中的螺旋参数测定(图1)。该方法首先通过最小旋转标架方法(rotation minimization frame,RMF)实现弯曲螺旋组装体的拉直,并将其沿着轴线展开,得到具有二维晶格结构的展开密度图;然后通过聚合多个不同半径的柱面信息,得到增强的衍射功率谱;最后利用功率谱中的衍射点位置确定倒易基向量并解算螺旋参数。除此之外,HELIS软件中还集成了螺旋纤维追踪、单纤维三维重构以及相对极性估计等功能,便于用户观察与分析样品中螺旋组装体的形态及相互关系。
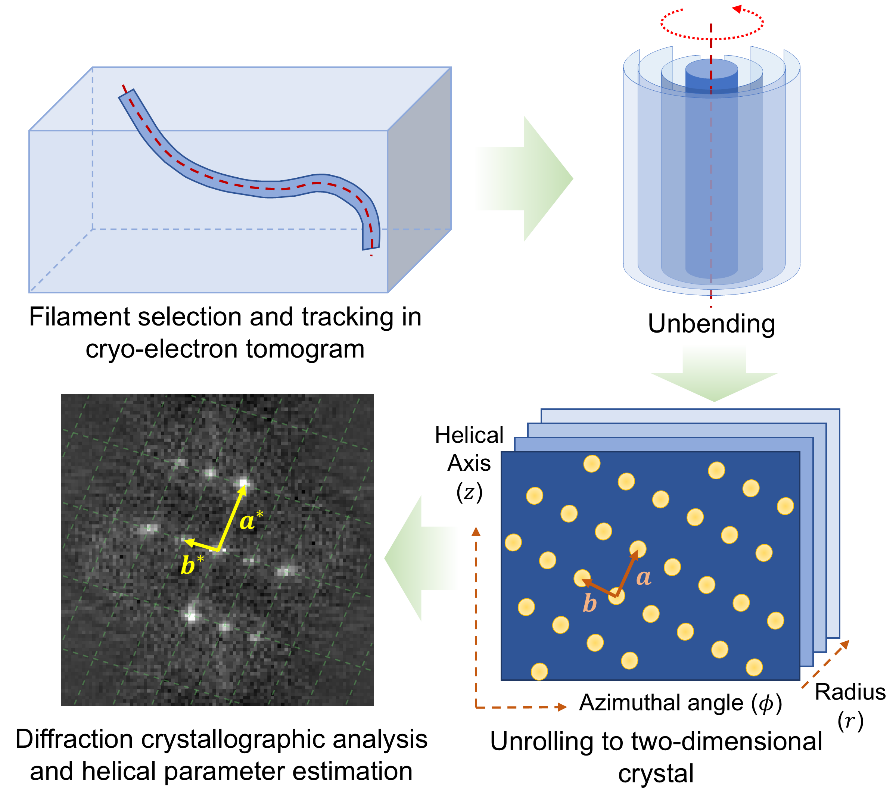
图1:基于柱面展开的螺旋参数估计方法流程图
研究团队使用了多种形态差异较大的螺旋组装体验证了方法的有效性与准确性。在光依赖型原叶绿素氧化还原酶(light-dependent protochlorophyllide oxidoreductase, LPOR)数据的测试中,HELIS顺利得到了清晰的衍射功率谱(图2c),实现了准确的螺旋参数估计,并通过对称平均展示了LPOR的每个对称单元都是二聚体(图2d)。对于拟菌病毒的基因组纤维(mimivirus genomic fiber, MGF)数据的测试展示了HELIS能够灵敏地检测出同种基本单元形成的若干种相近但不相同螺旋组装形式(图3)。在其他类型的数据上也展示出很高的准确性。相关的结果为高分辨结构解析以及结构功能研究奠定了必要的基础。
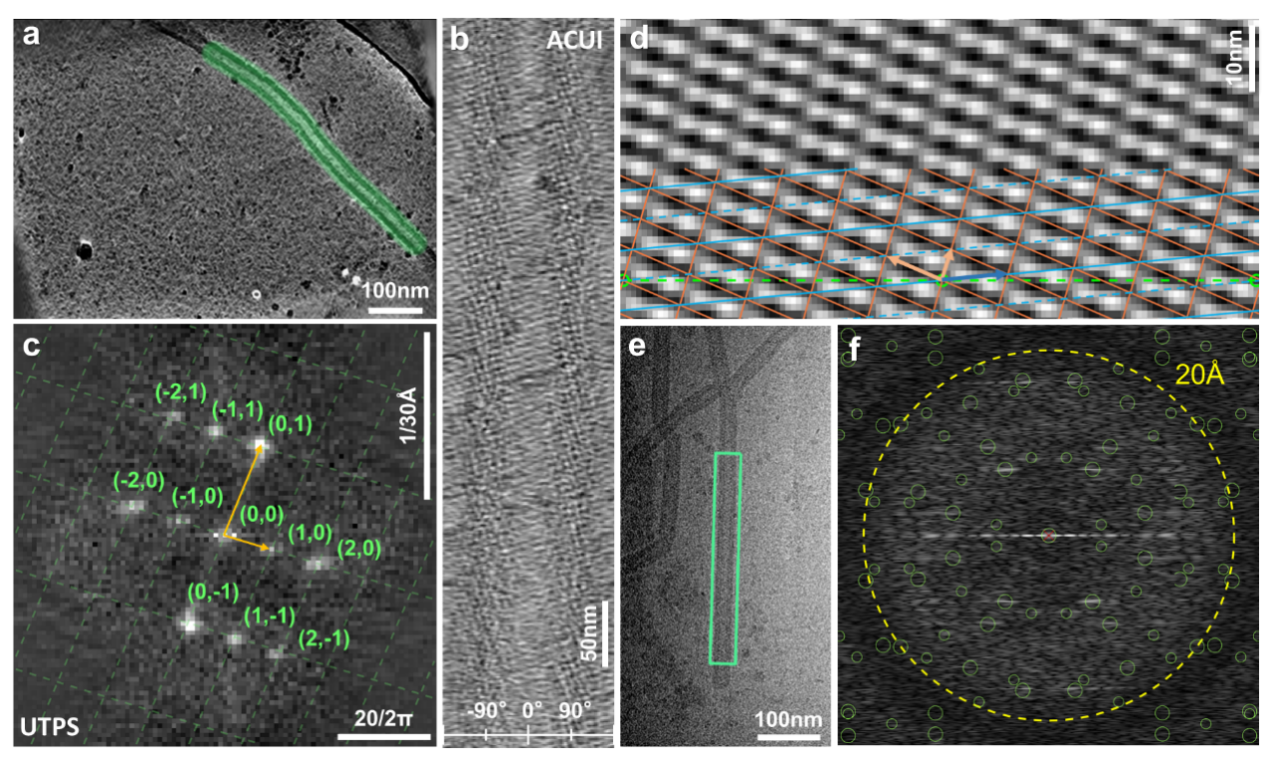
图2:LPOR膜管的实验结果。(a) 某根LPOR膜管的断层图像截面;(b) LPOR膜管的平均柱面展开图像;(c) LPOR膜管的管状展开功率谱。橙色箭头指示选定的一对倒易基向量,绿色数字表示衍射点的指标;(d) (b)的对称平均图像;(e) LPOR膜管的单颗粒图像;(f) LPOR膜管一段区域((e)中绿色框)的功率谱;在(b)和(c)中,水平方向和垂直方向的长度单位不同,两个方向分别提供比例尺。

图3:MGF螺旋异质性分析。(a)-(c) MGF三种不同螺旋组装体的平均柱面展开图像,下方嵌入图为叠加晶格的对称平均图,右上角显示GMC氧化还原酶二聚体的放大图;(d)-(f) 与(a)-(c)对应的管状展开功率谱,虚线表示倒易晶格;(g) 倒易晶格差异比较。在同一图像中用相应颜色的直线网格展示了(d)-(f)中的倒易晶格。
清华大学生命科学学院李雪明副教授和清华大学电子工程系沈渊教授为本文的共同通讯作者。清华大学生命科学学院和电子工程系2019级博士生黄铭涛为该论文的第一作者。本研究工作获得了国家自然科学基金委、生命科学联合中心、北京生物结构前沿研究中心等的资金支持,并得到了国家蛋白质科学研究(北京)设施清华基地的技术和设备支持。
论文链接:https://doi.org/10.1016/j.str.2025.06.008
软件链接:https://thuem.net/software/helis/overview.html