哺乳动物胚胎的着床前后转变(Pre- to post-implantation transition, PPT)是早期胚胎发育过程中的关键生物学事件。在这个发育窗口期,胚胎经历剧烈的发育重编程,在细胞和分子水平发生一系列精密调控事件,例如多能性状态的转变、第二次细胞命运决定、前后对称性打破、大规模的表观遗传重编程等。近年来,少量细胞或单细胞组学测序和分析技术的进展,极大推动了早期发育分子动态图景的绘制与理解,多个研究组曾报道哺乳动物胚胎在早期发育过程中DNA甲基化(DNA methylation,DNAme)的动态特征。作者团队曾报道人类着床后胚胎发育经历了谱系特异性DNA加甲基化1,然而该过程是否在物种间保守,以及哺乳动物胚胎PPT过程中DNAme和与基因表达(RNA expression,RNAex)如何协同调控、是否影响谱系命运转变,其机制有待进一步揭示。
2025年6月3日,清华大学生命科学学院、清华-北大生命联合中心周帆课题组在《细胞报告》(Cell Reports)杂志在线发表了题为“解析早期哺乳动物发育中DNA甲基化-转录的着床前后转变”(Dissecting pre- to post-implantation transition of DNA methylome-transcriptome dynamics in early mammalian development)的研究论文。该研究结合高精度多组学挖掘与体内外模型验证等体系,从围着床胚胎主要谱系及其亚群特异性DNA加甲基化模式、基因组元件上父母源DNAme差异、启动子DNAme与RNAex协同变化等多个角度,系统解析了围着床胚胎DNAme和RNAex协同动态调控模式。

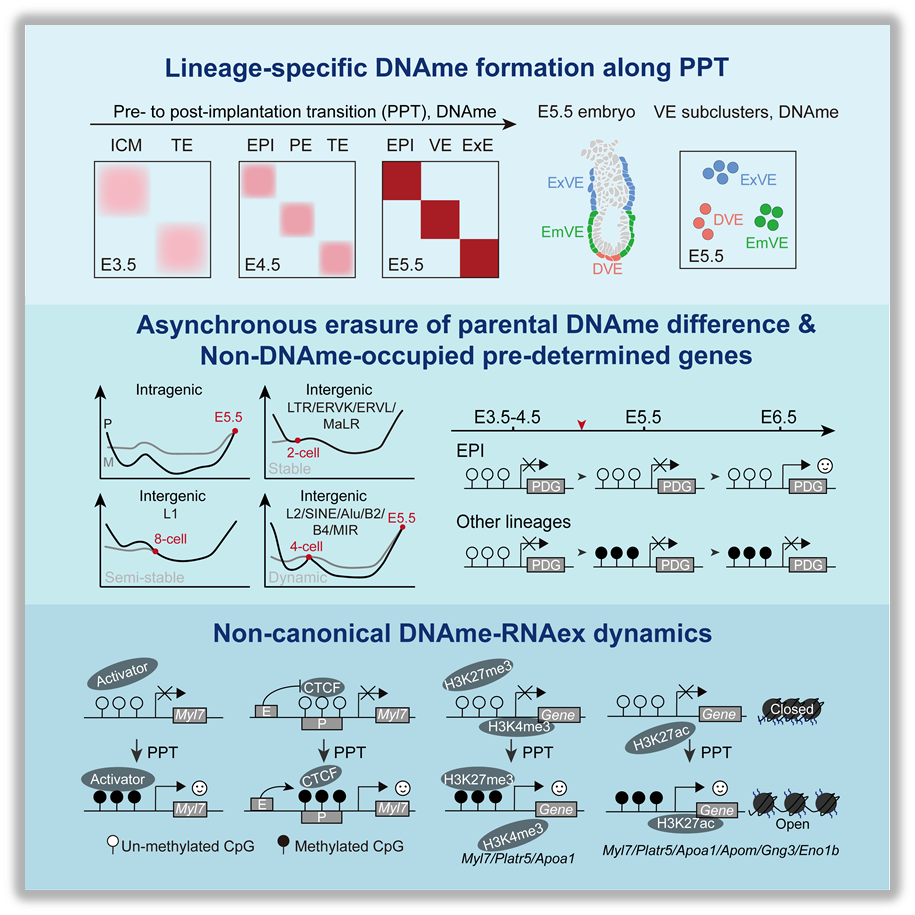
作者通过多组学测序中的RNA测序分别挑选出E3.5-E5.5小鼠胚胎细胞用于单细胞DNAme测序,结果显示着床后谱系均经历了全基因组甲基化水平的剧烈升高,其中胚内谱系(胚胎上胚层(epiblast,EPI))的DNAme重建速度与水平显著高于胚外谱系(内脏内胚层(visceral endoderm,VE)和胚外外胚层(extraembryonic ectoderm,ExE))。小鼠、猴和人类在受精后都经历了全基因组去甲基化过程,并在围着床阶段降至最低水平,着床后重新建立谱系特异的DNAme模式。此外,VE亚群在基因组元件和VE亚群特异表达基因上均呈现出差异DNAme,表明VE亚群命运决定过程中也经历了各自的DNAme动态变化(图1)。
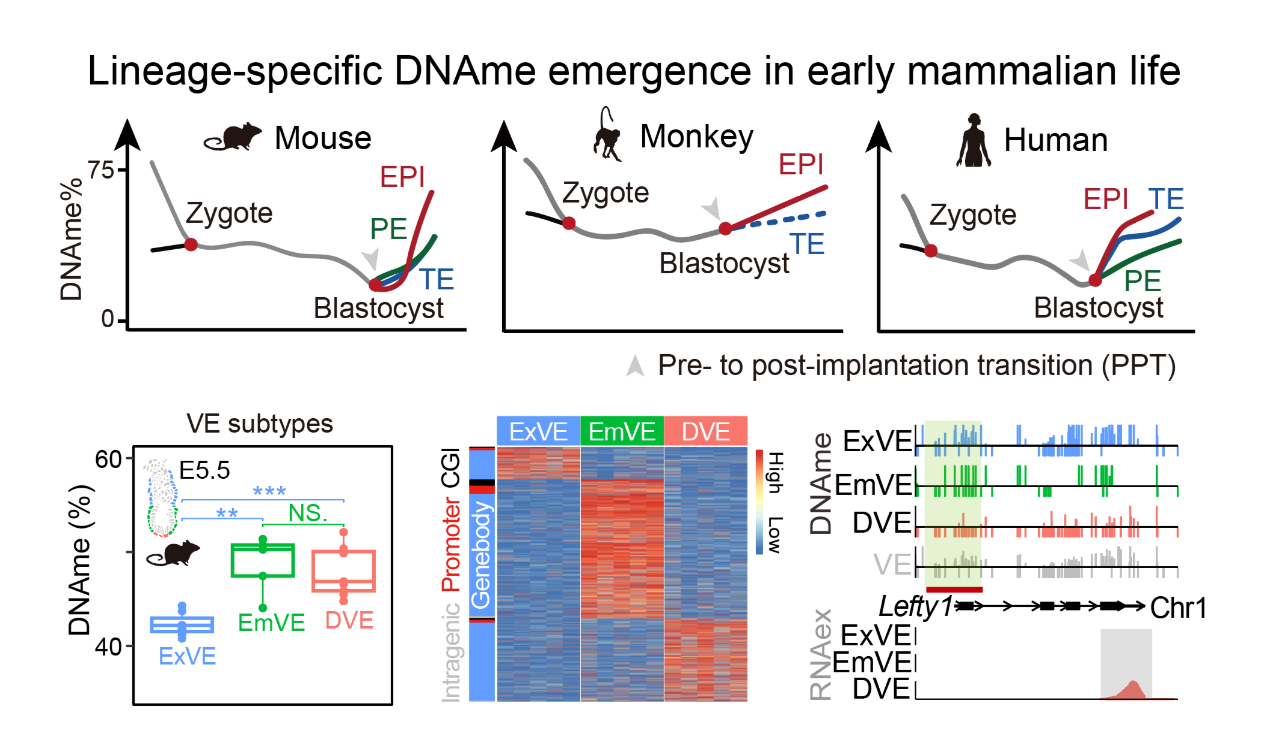
图1: 哺乳动物早期谱系特异性DNAme的出现
父母源差异性DNAme在印记基因的表达和转座元件的不对称沉默中发挥重要作用2-3,然而哺乳动物早期发育中该差异贯穿PPT全程的规律仍未系统揭示。作者发现早期胚胎发育过程中父母源DNAme差异的消失呈现出元件亚型特异性的时序差异,且这一现象与小鼠的性别和品系无关。值得注意的是,各类父母源基因组元件(尤其是转座子、逆转座子等区域)的异步动态变化,是否参与早期胚胎基因表达调控并影响谱系命运值得关注(图2)。
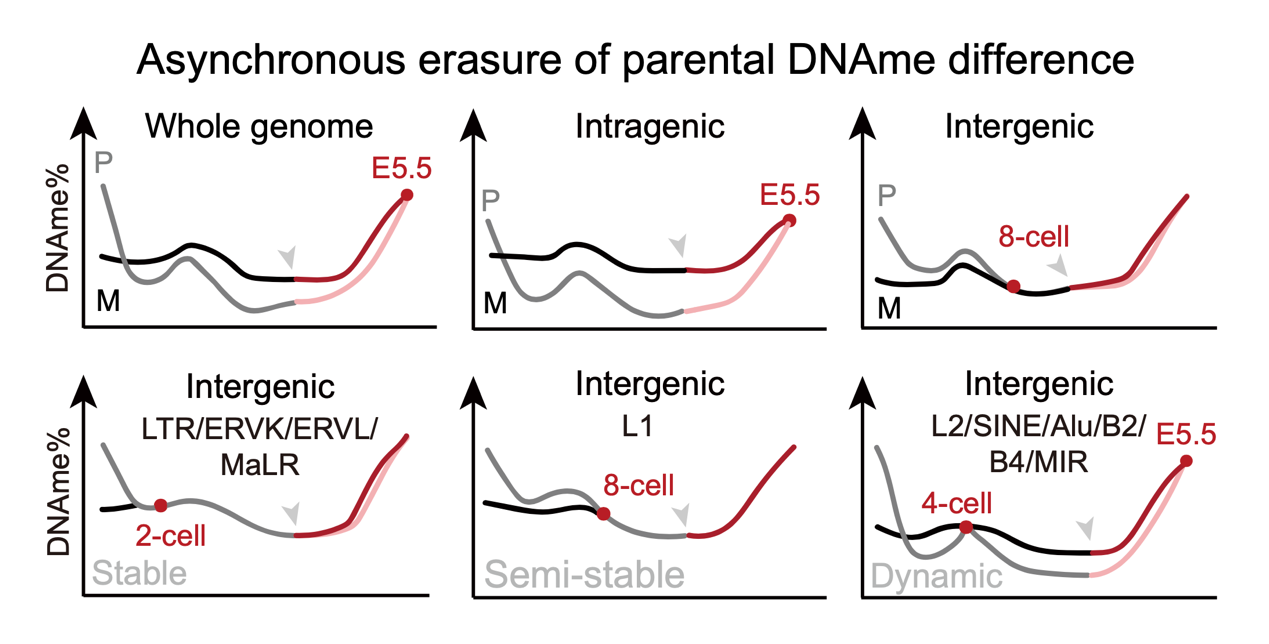
图2: 早期胚胎发育中父母源DNAme差异的异步擦除
启动子差异DNAme分析显示,E5.5胚胎存在EPI特异性低DNAme启动子,相关基因显著富集于E6.5原肠胚形成等关键发育通路。例如原肠运动相关基因Brachyury(T)在E5.5 EPI中提前形成EPI特异性低DNAme,并在E6.5原条(primitive streak, PS)中特异性激活表达。VE/ExE特异性低甲基化启动子相关基因主要参与调控消化系统与胎盘发育。这些谱系特异性启动子低甲基化基因与后续谱系特化和细胞命运决定密切相关,提示启动子甲基化状态可能通过"表观遗传预编程"机制预先调控各谱系基因开关,从而限制细胞向非目标谱系分化(图3)。
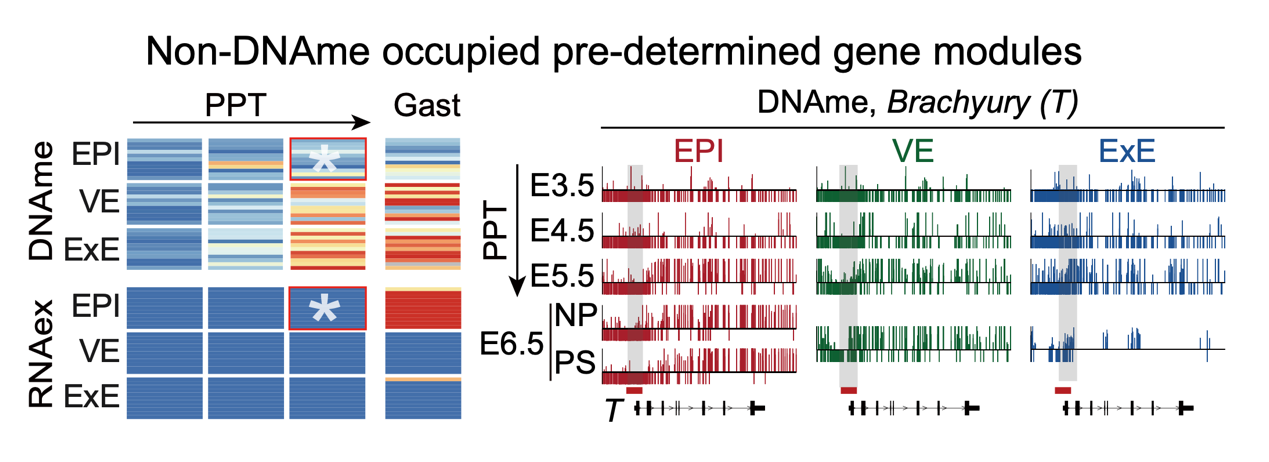
图3: 非DNA甲基化占据预设基因模块的建立
整合分析表明,围着床期EPI中大部分基因呈现经典DNAme-RNAex负相关模式(即启动子高甲基化抑制基因表达)。同时,少量基因存在启动子甲基化与RNAex正协同变化模式,且该模式在小鼠和人类各谱系中保守。通过过表达/敲除Dnmt3a/b的mESCs模型及分析Dnmt1/3a/3b三敲胚胎EPI,进一步验证此类非经典DNAme-RNAex正协同模式在干细胞和胚胎发育中的普适性。多维度调控因子分析提示,该表观遗传调控模式可能通过排斥CTCF、招募特异性激活因子(如Znf692)等多维分子机制共同调控谱系发育基因表达(图4)。
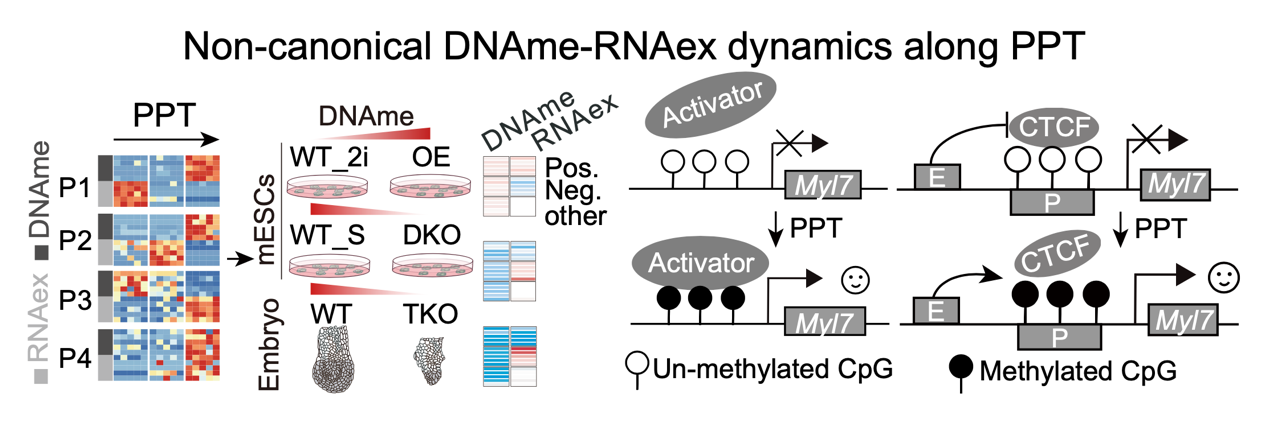
图4: 着床前后转变中的非经典DNA甲基化和转录动态的形成
总之,这项研究揭示了围着床胚胎谱系特异的DNAme动态变化及VE亚群DNAme差异模式;解析了不同基因组元件上父母源DNAme差异的非同步消失规律;鉴定了围着床期启动子DNAme-RNAex的多样化动态模式,特别发现一类DNAme-RNAex正协同变化的非经典分子特征,为理解胚胎发育中表观遗传与基因表达的互作关系提供了新视角。
清华大学生命科学学院周帆副教授为该论文的通讯作者。周帆课题组2023级博士生朱庆元和2021级博士生刘莹为并列第一作者,参与该工作的还有2023级博士生郝鑫、2020级博士生鄢胜镒、2020级博士生葛吉涛和2023级博士生郑长青。这项研究还得到了昌平国家实验室任仙文研究员的大力支持。清华大学颉伟教授及实验室成员卢绪坤、吴希和王利娟博士、北京大学季雄教授及实验室成员蒋永鹏博士、北京大学伊成器教授及实验室成员杨锦民博士生、中科院脑智卓越中心周昌阳研究员、清华大学孟安明教授及实验室成员张天奕博士生,哥伦比亚大学X. Shawn Liu教授,中科院生物物理研究所的朱冰研究员为本研究提供了实验试剂与技术、课题讨论等重要帮助和启发。本研究获得北京市自然科学基金、国家重点研发计划、细胞生态海河实验室创新基金、国家自然科学基金、清华大学自主科研计划、清华-北大生命科学联合中心、清华大学生命科学学院启动经费、膜生物学全国重点实验室等项目的资助。该研究还得到清华大学实验动物中心,生物计算平台和膜生物学全国重点实验室平台的支持与帮助。
原文链接:https://www.cell.com/cell-reports/fulltext/S2211-1247(25)00561-3
1. Zhou, F., Wang, R., Yuan, P., Ren, Y., Mao, Y., Li, R., Lian, Y., Li, J., Wen, L., Yan, L., et al. (2019). Reconstituting the transcriptome and DNA methylome landscapes of human implantation. Nature 572, 660-664. 10.1038/s41586-019-1500-0.
2. Guo, F., Li, L., Li, J., Wu, X., Hu, B., Zhu, P., Wen, L., and Tang, F. (2017). Single-cell multi-omics sequencing of mouse early embryos and embryonic stem cells. Cell Res 27, 967-988. 10.1038/cr.2017.82.
3. Smith, Z.D., and Meissner, A. (2013). DNA methylation: roles in mammalian development. Nat Rev Genet 14, 204-220. 10.1038/nrg3354.