凭借着序列互补这一独特的性质,核酸分子在众多生物大分子当中脱颖而出,成为合成生物学中的研究热点之一。以DNA为例,DNA分子中的四种组成A、T、C、G之间有着严格的匹配规则——Watson-Crick碱基互补配对(A与T配对,C与G配对)。这种碱基之间的匹配规则进而造成了碱基序列与碱基序列之间的特异性识别,即序列互补。对于任意一条DNA单链而言,其互补链的序列是明确且唯一的;错误匹配的序列会使降低两条链之间的结合速率,并使得最终形成的双螺旋结构的稳定性大大降低。基于DNA序列间的特异性识别,科研人员已经大大拓展了对DNA的研究,不再单一地将其作为遗传信息的载体,更是将其用作分子探针和分子机器人,使DNA分子在诸如药物递送、生物计算、疾病诊断、超分辨成像等众多领域显现出极其可观的应用前景。与此同时,在DNA纳米结构设计的不断探索中,人们对结构的复杂性、灵活性及功能化等方面的认识愈渐深入, 相关设计理念和设计经验得以不断的积累和丰富。
在DNA纳米结构领域中,研究人员一般通过设计粘性末端(sticky ends)来实现结构之间的识别。这些人工设计的粘性末端有着特定的长度和序列,只有序列互补的两个粘性末端之间才会形成完美的识别,从而引导DNA纳米结构的静态或动态组装。理论上长度为N(碱基数)的粘性末端可以形成4N种序列 。但是在实际设计中,粘性末端的长度和序列通常会受到一定的限制,使得最终的特异性序列的种类大大指数性降低。例如对四种碱基来说,长度为2 碱基的粘性末端只能形成42=16种互补对,长度为1 碱基的粘性末端只能形成41=4种互补对。如何在实际设计中优化和提升粘性末端之间的识别能力已经成为DNA分子研究的一大重点。
在本研究中,作者从中国传统木工艺的榫卯结构中获得灵感,设计了全新的DNA粘性末端之间的识别方式——依赖几何构型的特异性识别。与依赖序列互补配对的识别方式不同,这种识别方式完全不依赖于序列的多样性,而是基于DNA双螺旋结构的多种几何构型之间的形状契合而实现的。作者展示了即便在最极端的设计情景——粘性末端长度仅为1碱基且碱基种类只有C-G配对的情况下,依然可以实现将仅有的1种互补对提升为至少10种互补对,显著地提高了DNA粘性末端之间的识别能力。
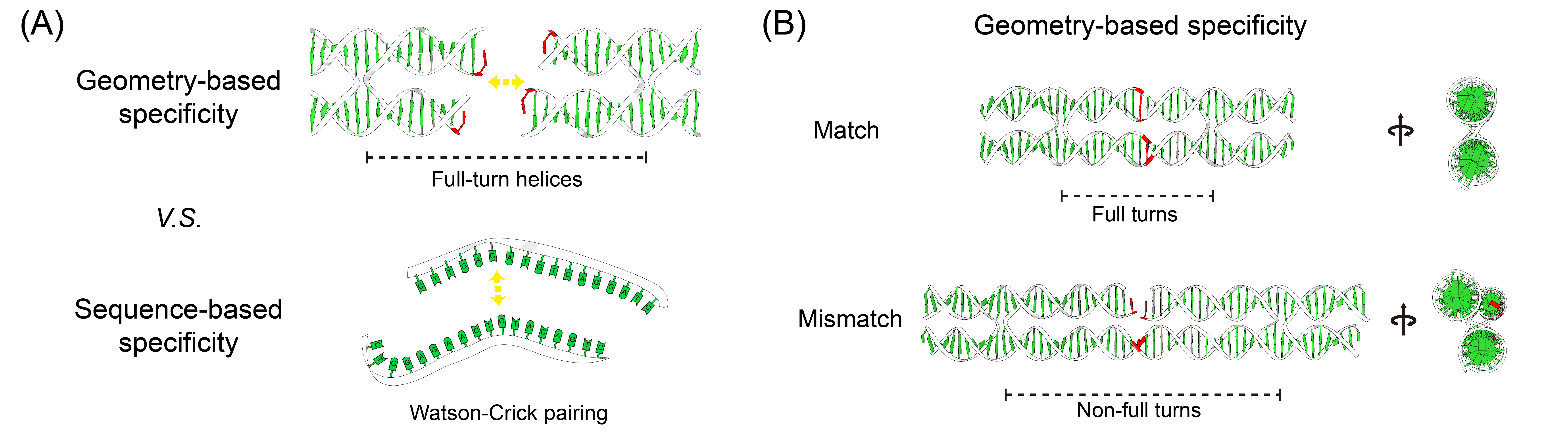
图 1. 基于几何构型的DNA分子特异性识别。 (A)依赖几何构型的特异性识别方式与依赖于序列互补的特异性识别方式之间的对比。(B)在依赖几何构型的特异性识别方式中,只有当两个粘性末端之间满足特定的几何构型要求时才会发生结合;当两个粘性末端之间没有满足特定的几何构型要求时,不会发生结合。
作者以简单的四臂分支型DNA纳米结构为模板,对其分支处的粘性末端进行设计优化,通过插入特定数量的碱基对使粘性末端的成键界面呈现出多种不同的几何构型。只有当两个末端相连使整体的几何构型满足B型DNA的螺旋参数时,才会发生结合,否则这两个末端不能发生稳定结合。利用四臂分支型DNA纳米结构的二维自组装结果来验证和分析依赖几何构型的特异性识别方式的可行性。随后利用这种识别方式成功地实现了与设计预期严格相符的多种多样的二维及三维组装。
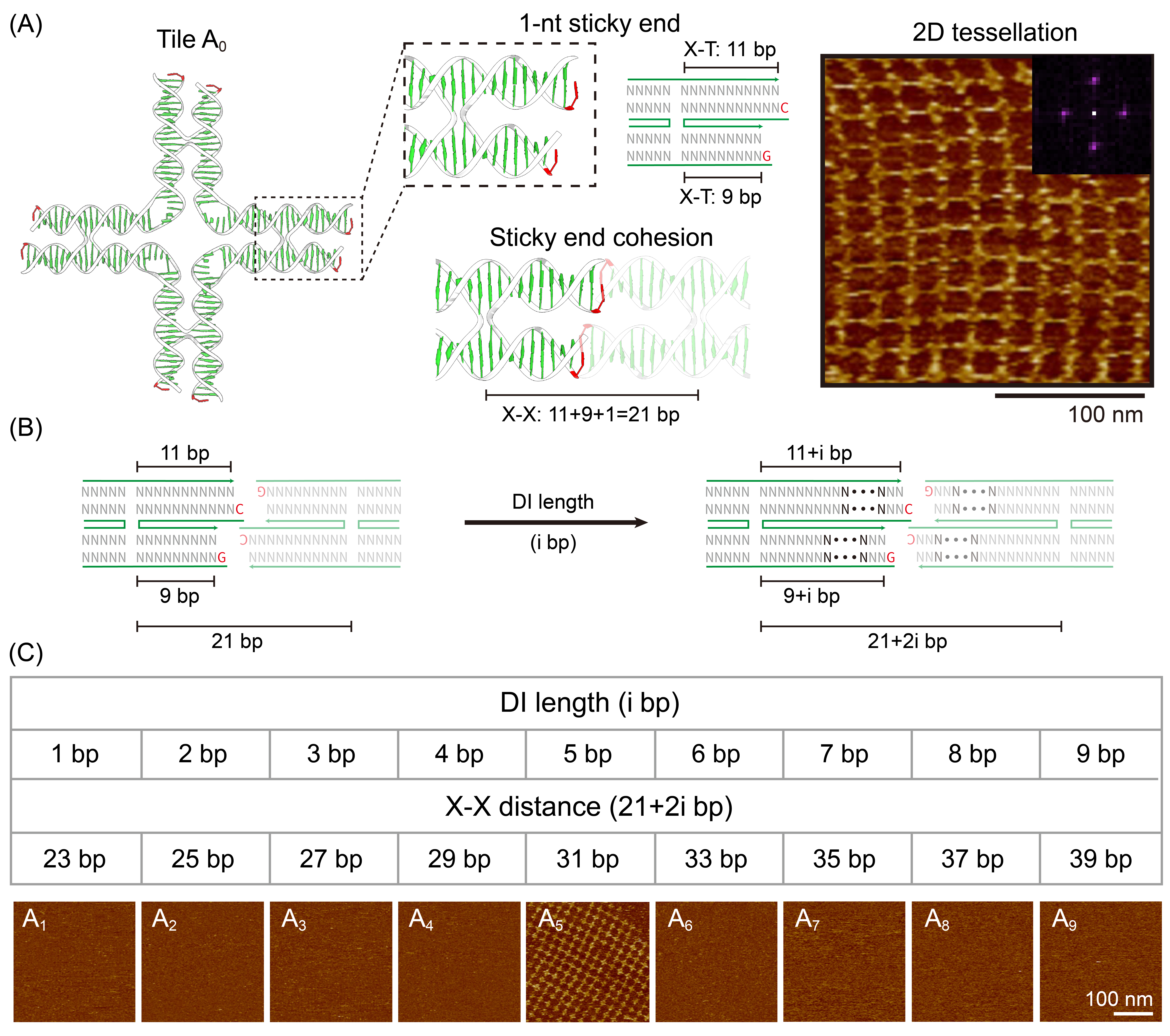
图 2. 在四臂分支型DNA纳米结构中应用依赖几何构型的DNA粘性末端。(A)四臂分支型DNA纳米结构的设计细节及二维自组装活性。(B)依赖几何构型的DNA粘性末端的设计细节。(C) 粘性末端的成键界面呈现出多种不同的几何构型,展现出不同的组装活性。只有当两个末端相连使整体的几何构型满足B型DNA的螺旋参数时,才会发生二维组装。
本研究拓展了DNA粘性末端的识别方式,建立了新的成键机制和调控方法,提升了DNA分子之间的识别能力,对今后高灵敏度、高精度DNA分子探针及分子机器的设计和应用具有重要的意义。
该研究成果由清华大学生命学院魏迪明分子设计课题组(MADlab)完成,论文题为“基于分子之间的几何构型识别方式设计特异的DNA粘性末端”(Design of orthogonal DNA sticky end cohesion based on configuration-specific molecular recognition),于2022年9月30日发表于《美国化学学会志》(Journal of the American Chemical Society)。清华大学生命科学学院2017级博士生张天晴为本文的第一作者及共同通讯作者,从项目构思、具体实施到最后论文撰写其全程基本独立完成,另一共同通讯作者为清华大学生命科学学院魏迪明副教授,在项目立意和写作方面提供了协助。该研究得到科技部、清华-北大生命科学联合中心等基金资助。
论文链接:https://pubs.acs.org/doi/10.1021/jacs.2c07181