想必大家都玩过经典游戏俄罗斯方块,有没有想过这些方块也可以在纳米尺度制备出来?DNA自组装和DNA计算就可用于实现这个奇异的想法。DNA自组装形成的复杂纳米结构和装置作为合成生物学元件,能够在生命科学、化学、材料学等领域中提供微纳米技术解决方案。在DNA纳米技术领域中,有许多的方法能够实现DNA纳米结构的动态变构,包括调节环境因素实现不同DNA结构状态的转换、利用酶对DNA分子的作用实现结构变换,以及经典的黏性末端链置换方法等等。许多的DNA动态系统和装置被成功构建。
本研究中提出用一种更简便、有效的基于非黏性末端链置换方法的系统,实现DNA纳米结构的动态转换。在本系统中,封闭链(blockers)能够通过竞争置换将已经形成互补配对的DNA链分离,进而实现简单的DNA结构和复杂的二维、三维DNA纳米结构的变换。通过调节封闭链和被竞争结构的浓度比,可有效地实现DNA结构状态的可逆转换。基于概率模型和反应平衡模型,计算机模拟揭示了该体系与传统方法相比的区别和优势。可逆的动态变构反应和简便的反应方向控制是本方法的显著特点。为了进一步应用在复杂的DNA结构中,本研究采用了二维长方形DNA纳米结构和三维长方体DNA纳米结构用于结构变换。在二维体系中,随着结构之间的反应位点从单一位点到多位点发展,结构之间的相互作用呈现协同效应,非黏性末端链置换方法仍然可以发挥转换结构的作用。在三维体系中,非黏性末端链置换不仅体现出其对于结构变换的控制性能,还可以用于构建布尔函数(Boolean functions)。本研究中利用两个单元和四个单元的三维长方体DNA纳米结构分别构建出了4比特输入/2比特输出和16比特输入/8比特输出的多入多出布尔函数(MIMO Boolean functions)。如图1所示,通过封闭链的控制,四种基本三维长方体DNA纳米结构可以形成七种不同的俄罗斯方块。
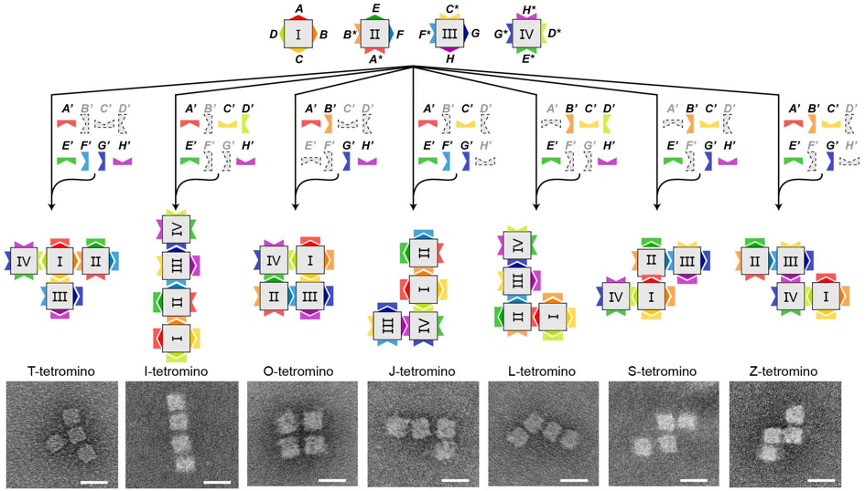
图1 基于非黏性末端链置换方法的三维DNA长方体纳米结构单元形成不同的四聚体复合体的操作和结果。四种三维长方体DNA纳米结构单元的四个侧面分别相互配对(相互匹配的颜色的凹凸面代表可以配对的侧面和可配对的封闭链)。当特定的封闭链集合(用彩色表示使用的封闭链,用灰色表示未采用的封闭链)加入到相同的四种单元混合体系中,能够控制形成七种俄罗斯方块中的一种。最下排为电子显微镜成像结果,比例尺:25纳米。
在计算过程中,由于三维DNA长方体纳米结构的外形易于识别,可以用于表示特定系统的状态,因而本研究中将结构的形状信息作为布尔函数运算的输出。进行多入多出布尔函数计算时,可以通过函数之间的组合和迭代,通过不同反应方式得到相同的结果,即用不同的运算过程得到相同的输出,见图2。能够实现在DNA计算过程中观测中间结果,同时也为DNA计算提供了一种条件控制的方法。
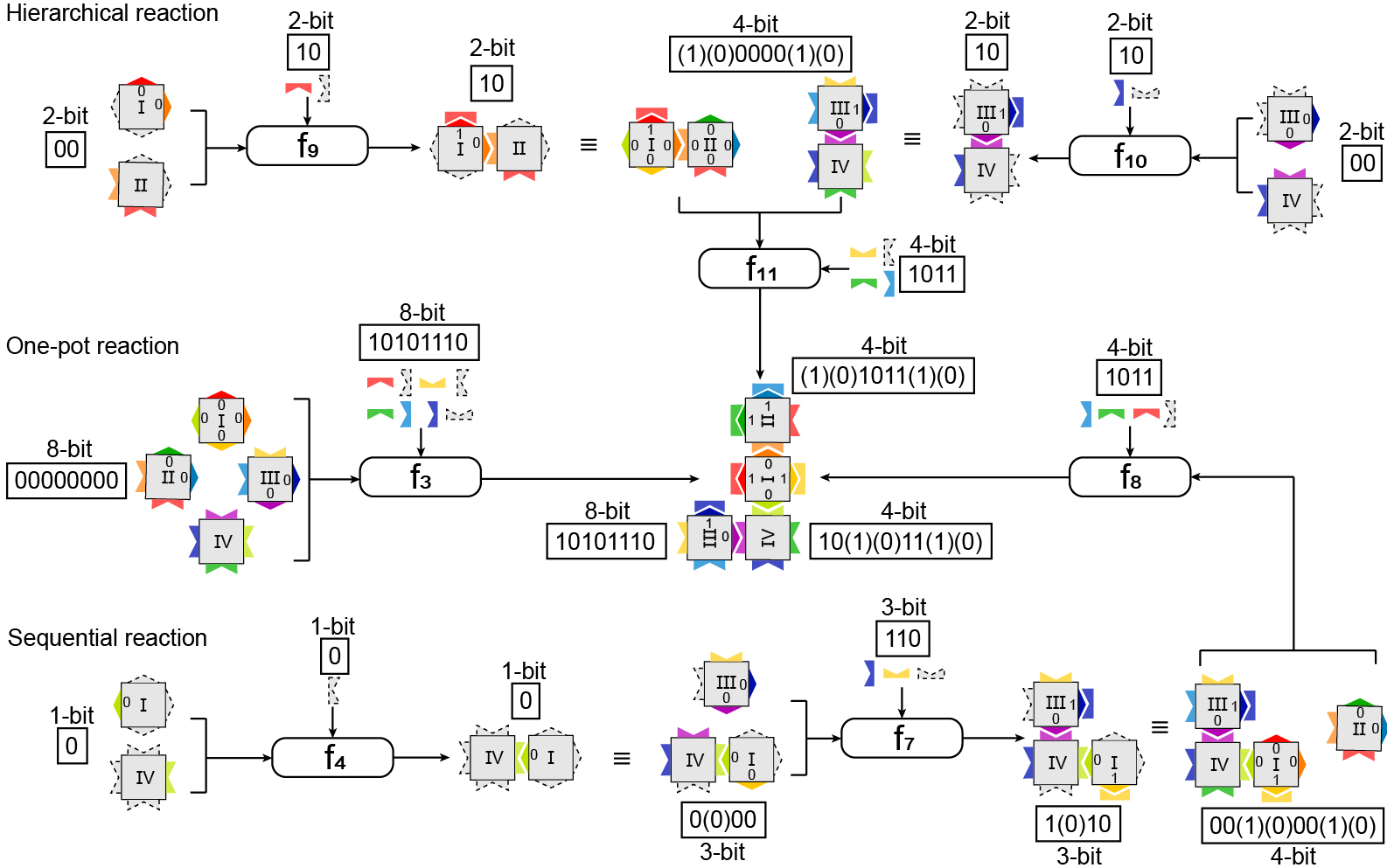
图2 基于非黏性末端链置换方法控制信息计算过程。采用三种不同方式反应可以得到相同的J型四聚体俄罗斯方块。第一种是层级式反应(顶部)。两两单元之间形成二聚体,然后再形成四聚体,即两个2比特输入/2比特输出的布尔函数通过组合得到一个4比特输入/2比特输出的函数。第二中是一步法反应(中间),操作与图1所示相同,是一个16比特输入/8比特函数输出的布尔函数。第三种是顺序反应(底部),单元之间依次连接形成四聚体,是三个2比特输入/2比特输出布尔函数之间的迭代。
该研究成果的论文题为“非黏性末端链置换方法在DNA动态和计算中的研究”(DNA dynamics and computation based on toehold-free strand displacement),由清华大学生命学院魏迪明分子设计课题组(MADlab)于2021年8月17日发表于《自然通讯》(Nature Communications)期刊上。
多学科深度交叉是此研究的一个鲜明特点,清华大学生命科学学院博士生康宏为本文的第一作者,共同通讯作者清华大学生命科学学院魏迪明副教授提出概念和实验体系,共同通讯作者清华大学自动化系的贾庆山教授在布尔计算方面做出了重要贡献,另外一名共同通讯作者香港科技大学的Lakerveld副教授在计算机模拟方面也做出了不可或缺的贡献。该研究得到国家自然科学基金委、科技部、教育部、清华-北大生命科学联合中心、清华大学结构生物学高精尖中心等基金资助。
原文链接:https://doi.org/10.1038/s41467-021-25270-7