近日, 清华大学生命学院方显杨课题组与美国南加州大学化学系Peter Z. Qin教授课题组合作在《化学科学》(Chemical Science)杂志发表了题为“基于非天然碱基对系统的非变性条件下长链RNA转录后位点特异性自旋标记技术”(Posttranscriptional site-directed spin labeling of large RNAs with an unnatural base pair system under non-denaturing conditions)的文章。
电子顺磁共振技术(Electron Paramagnetic Resonance, EPR)与核磁共振技术(Nuclear Magnetic Resonance, NMR)类似,都是磁共振波谱学技术的分支。近年来,位点特异性自旋标记电子顺磁共振技术(Site-Directed Spin Labeling Electron Paramagnetic Resonance, SDSL-EPR)已成为研究生物大分子(包括蛋白质, DNA, RNA等)的结构、动态特性与功能的重要技术。这项技术能够在近生理条件下探测生物大分子不同功能状态下的动态特性信息、提供自旋中心间20-100埃的距离信息、并且实时的检测生物大分子的构象变化过程等。SDSL-EPR具有灵敏度高(相比于核磁共振), 对生物大分子的分子量和均一性均没有限制的优点,SDSL-EPR已日益成为重要的整合结构生物学研究工具。
由于大多数生物大分子是天然逆磁性的,开展SDSL-EPR研究的前提是向生物大分子特定位点引入一个或成对的自旋标记,这些自旋标记可以是自由基或顺磁金属离子。当前,蛋白质的位点特异性自旋标记技术已有很好的发展而相对成熟,而RNA,特别是长链RNA的位点特异性自旋标记仍十分具有挑战性。例如,基于固相化学合成的标记方法主要局限于长度在100个核苷酸以下的短链RNA;基于DNA夹板指导的酶促连接法可实现长链RNA的位点特异性自旋标记,但其步骤往往十分繁琐,还需要经历变性复性过程,该方法不仅效率低,其变性过程常常导致长链RNA的不可逆错误折叠。
针对现有RNA位点特异性自旋标记技术的局限性,清华大学生命学院方显杨课题组利用包含NaM-TPT3非天然碱基对的系统,通过化学合成碱基带有炔基修饰的TPT3(rTPT3CO)(图1A),经过PCR和体外转录反应,在RNA中位点特异性的引入TPT3CO,进一步利用点击化学反应的高选择性,通过与叠氮基修饰的氮氧自由基化合物反应,建立了在非变性条件下长链RNA位点特异性自旋标记的策略,并被成功应用于长度为419核苷酸的核糖核酸酶RNAse P的位点特异性自旋标记中(图1B)。通过与美国南加州大学化学系Peter Z. Qin课题组合作,应用连续波(CW)EPR 技术证实了该方法的高标记效率、 研究了标记位点附近的动态特性、以及用双电子自旋共振(DEER) EPR技术测定了成对自旋中心间的距离(图1C)。该标记策略的成功建立将促进SDSL-EPR技术在长链RNA的结构与动态特性研究中的应用,并推动建立长链RNA的整合结构生物学研究方案。
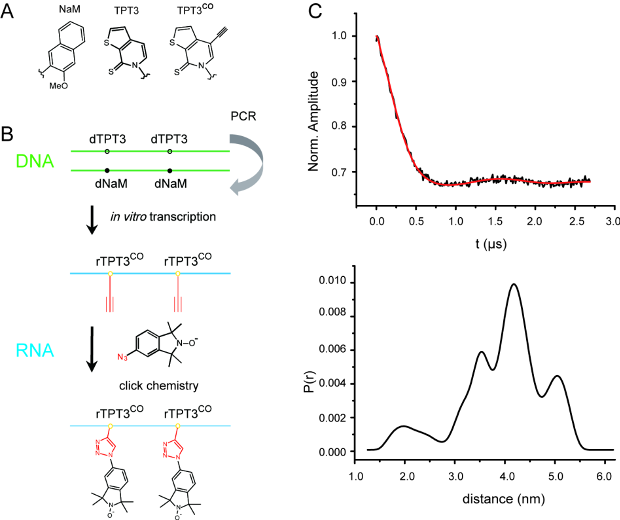
图1.基于非天然碱基对系统的长链RNA位点特异性自旋标记方案及其在电子顺磁共振中的应用。
清华大学生命学院方显杨研究员与美国南加州大学化学系Peter Z. Qin教授为该文的共同通讯作者。清华大学2016级PTN直博生王岩为该文的第一作者。南加州大学化学系博士后Venkatesan Kathiresan、博士生Wei Jiang参与了DEER EPR数据采集;清华大学生命学院2014级本科生陈垚宜(现在德国Freie Universität攻读博士学位)、2017级CLS直博生胡艳萍参与了研究工作;北京大学药学院化学生物系刘国全研究员、博士生白光灿在CW EPR数据收集方面提供了大力帮助。该项目受到国家自然科学基金委大科学装置科学研究联合基金重点支持项目、北京结构生物学高精尖创新中心、清华-北大生命科学联合中心的经费支持。
原文链接:https://doi.org/10.1039/D0SC01717E