2018年1月9日,清华大学生命科学学院陈春来研究组在《Cell Reports》杂志发表了研究论文,题目为“The Conformational Dynamics of Cas9 Governing DNA Cleavage Are Revealed by Single-Molecule FRET”(利用单分子FRET揭示Cas9在DNA切割过程中的动态构象)。该论文展示了Cas9可以自发地在三种主要构象中转换,揭示了Cas9蛋白与PAM远端DNA/RNA双链相互作用可长程别构调控Cas9切割结构域的局部构象,这是Cas9切割靶标DNA之前的最后校验步骤。最后,该文提出了优化和设计高保真Cas9的新思路,即通过突变Cas9蛋白以影响这种长程别构调控机制,可提高Cas9的识别特异性。
CIRSPR/Cas9是一种可以由一条单链sgRNA(single guide RNA)指导的利用Cas9核酸酶对靶向DNA进行特异性识别和切割的技术。由于其可以高效快捷地靶向切割DNA而被广泛地应用于基因编辑、基因表达调控、基因定位和成像等领域。但是,Cas9的脱靶效应严重阻碍了其应用。尽管有一些研究报道了通过改造Cas9和截短sgRNA来提高其特异性,但仍然缺乏对Cas9切割靶标DNA的详细分子机制的全面认知,因而难以对CIRSPR/Cas9的优化提供系统性的指导。
单分子荧光共振能量转移技术(FRET)是检测生物大分子构象变化的有利工具。该研究通过在Cas9蛋白的特异位点上标记荧光团,基于全内反射荧光显微镜快速灵敏地捕捉单个Cas9分子的FRET效率变化以及在每个FRET状态的停留时间,以此反应Cas9的构象变化,并提供该过程中的一系列动力学参数。该研究发现,Cas9展现出三种构象状态,并可以自发的在这三种状态中自由切换。靶标序列的PAM远端与sgRNA的大于三个碱基的错配,导致Cas9的HNH结构域(核酸切割结构域)稍偏离了其正确切割位点,因而将其从切割活性态转化为非活性状态。而这种对错配的校验机制是Cas9蛋白与PAM远端DNA/RNA双链相互作用对其HNH结构域的长程别构调控来实现的。
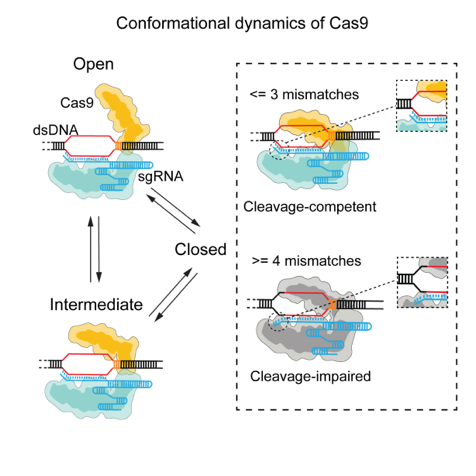
图1. Cas9蛋白切割靶标DNA的动态动态构象和校验机制模型
通过对Cas9一些高保真突变体的研究,该文章证明了在Cas9/RNA/DNA三聚复合物中引入额外的能量损耗导致Cas9切割底物的能垒升高,从而提高了Cas9切割特异性。该文章为优化Cas9特异性提供了新思路,即通过减弱Cas9与PAM远端DNA/RNA异源双链的相互作用来提高切割能垒,从而达到提高特异性的目的。
清华大学生命科学学院陈春来研究员为本文通讯作者。清华大学生命学院CLS项目三年级博士生杨梦铱,清华大学生命学院三年级直博生彭思佳为共同第一作者。本工作获得了北京结构生物学高精尖创新中心、清华-北大生命科学联合中心及国家自然科学基金委的经费支持。
原文链接
http://www.cell.com/cell-reports/fulltext/S2211-1247(17)31872-7