2018年11月29日,清华大学生命科学学院植物生物学研究中心戚益军研究组在《自然通讯》(Nature Communications) 杂志在线发表了题为《拟南芥中长非编码RNA的系统鉴定揭示反义RNA调控MAF4基因》(Global identification of Arabidopsis lncRNAs reveals the regulation of MAF4 by a natural antisense RNA)的研究论文。该研究系统鉴定和分析了拟南芥中大量长非编RNA (long noncoding RNA,lncRNA),发现一个可调节开花时间的NAT-lncRNA,并阐明了其正向调控正义链基因转录的作用机制。
近年来,全基因组和转录组测序结果表明大约75%的人类基因组和50%的拟南芥基因组可转录成RNA,但蛋白编码序列只约占其中的1.5%。大量不能编码蛋白的 RNA进入人们的视野,其中包括数以万计的lncRNA。LncRNA为一类长度大于 200 个核苷酸、 不具备蛋白编码功能的 RNA。根据与蛋白编码基因的位置关系,lncRNA可以分为源于基因反义链的 lncRNA (NAT-lncRNA),与基因序列同向重叠的 lncRNA (OT-lncRNA),产生于基因间区的lncRNA (lincRNA) 以及来自于基因内含子区域的 lncRNA (incRNA) 等。越来越多证据表明,lncRNA 通过多种方式参与基因表达调控、染色体高级结构形成、RNA加工亚细胞结构组装等重要生命活动过程 。
在这项研究中,为了研究植物中 lncRNA对基因表达的调控功能,戚益军研究组利用链特异性 RNA-seq 结合生物信息学分析方法,系统鉴定和分析了模式植物拟南芥中的 lncRNA。该研究共鉴定到了6510 个 lncRNA ,其中包括 4050个 NAT-lncRNA 以及2460个 lincRNA。研究发现,在不同组织或逆境处理条件下,大量 NAT-lncRNA 与邻近蛋白编码基因的表达呈现正相关趋势。此外,通过人工 miRNA 的技术,沉默部分 NAT-lncRNA 的表达,可导致邻近蛋白编码基因表达下降,表明 NAT-lncRNA 可正向调控邻近基因的表达。该研究为解读植物中 lncRNA 的功能和作用机制提供了丰富的资源和良好的基础;MAS 正向调控 MAF4 转录的机制解析对研究植物中大量 NAT-lncRNA 的功能机制有重要的借鉴意义。
清华大学生命学院博士生赵新玥、连璧和现医学院博士后李景睿为该论文的共同第一作者。戚益军教授和李艳博士为论文共同通讯作者。该研究由国家自然科学基金委和清华-北大生命科学联合中心提供经费支持。
论文链接:https://www.nature.com/articles/s41467-018-07500-7
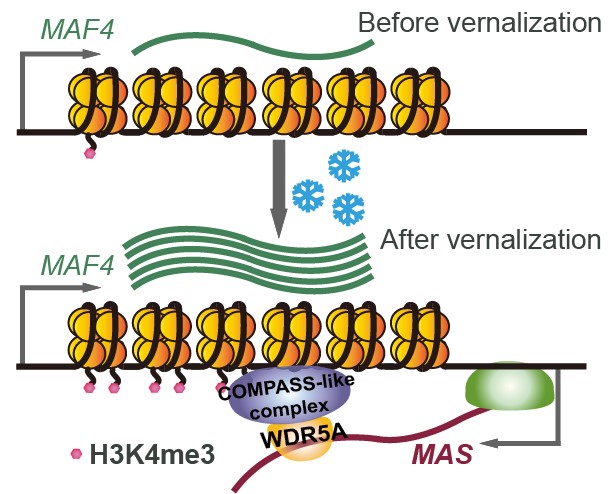
图1,MAS调控MAF4的作用机制