2020年3月27日,《分子细胞》杂志在线发表了题为“Drosha识别pri-miRNA的结构基础”(Structural Basis For pri-miRNA Recognition by Drosha)的研究论文,报道了中国科学院生物物理研究所许瑞明研究组和清华大学王宏伟课题组最新合作研究成果,该研究成果解析了Drosha-DGCR8复合物和pri-miRNA结合,对其进行加工的结构。
真核细胞中存在着大量不能翻译成蛋白质的RNA,被称为非编码RNA。它们在有机体基因表达调控等诸多方面发挥重要作用,广泛参与生长发育、组织分化等生物学过程,并且与疾病的发生、发展密切相关。microRNA(miRNA)是一类非常重要的非编码RNA,长度约为21~24个碱基。它们通过碱基互配与靶标mRNA相结合,从而抑制蛋白质翻译,或促进mRNA降解。在哺乳动物中,初始miRNA(pri-miRNA)伴随着基因组DNA转录而产生,其长度可达数千个碱基。其核心部分为一个茎环结构,功能miRNA包藏在该结构的茎部双链区。从pri-miRNA到成熟的功能miRNA需要依次经历发生在细胞核内与胞质中的两步切割反应,分别由III型RNA酶家族成员Drosha (核内)和Dicer(胞质)催化完成。2018年,王宏伟课题组解析了Dicer复合物与底物的冷冻电镜结构,阐明了Dicer对miRNA的识别加工机制。不同于Dicer识别底物的模式,Drosha发挥功能需要与DGCR8形成复合物(也被称为Microprocessor复合物),其中Drosha是核心催化组分,DGCR8为双链RNA结合蛋白,负责招募pri-miRNA底物。Drosha/DGCR8复合物早在2004年即被发现,然而一直以来,受限于难以获得高表达及稳定均一的重组蛋白等因素,其结构生物学研究进展缓慢。尽管前期的体外功能研究积累了大量的数据,包括鉴定了pri-miRNA上的关键序列、筛选了蛋白质的功能结构域等,但是并未解答这些序列究竟如何被识别、以及蛋白质功能域如何参与界定pri-miRNA切割位点这些重要科学问题。
在这项研究中,研究人员成功纯化、组装了人源Drosha/DGCR8,及其结合pri-miRNA的复合物。经过一系列条件优化,包括交联剂筛选、金属离子置换以稳定复合物结构,经过上千次电镜以摸索最合适条件,最后对之进行了结构解析。该结构证实了Drosha在切割位点界定中的决定性作用,并首次明确了PAZ、MB helix、以及dsRBD结构域分别对pri-miRNA的相应区域进行识别,协同完成切割位点的定位。其中,PAZ结构域在RNA结合前后出现了明显的构象变化,它与MB helix一起,结合在pri-miRNA的双-单链交界区的两侧,形成三明治状稳定结构。这个现象一方面从机制上阐明了pri-miRNA双-单链交界区是决定切割位点的关键顺式元件的原因,另一方面提出了PAZ结构域识别RNA的新模式。这项工作同时还发现了PAZ结构域的构象变化反映了Drosha的不同活性状态。在没有pri-miRNA结合时,PAZ的发卡状双螺旋占据了切割活性中心区域,从而阻碍了RNA的结合;当识别pri-miRNA底物时,该螺旋结构向下翻转并结合RNA,反而稳定了与底物的相互作用。研究人员认为,这是一种从自抑制状态到活化状态的转变,表明Drosha存在活性调控模式。由于体内存在很多miRNA类似物,这种可调控模式对于底物的特异性识别具有重要意义。
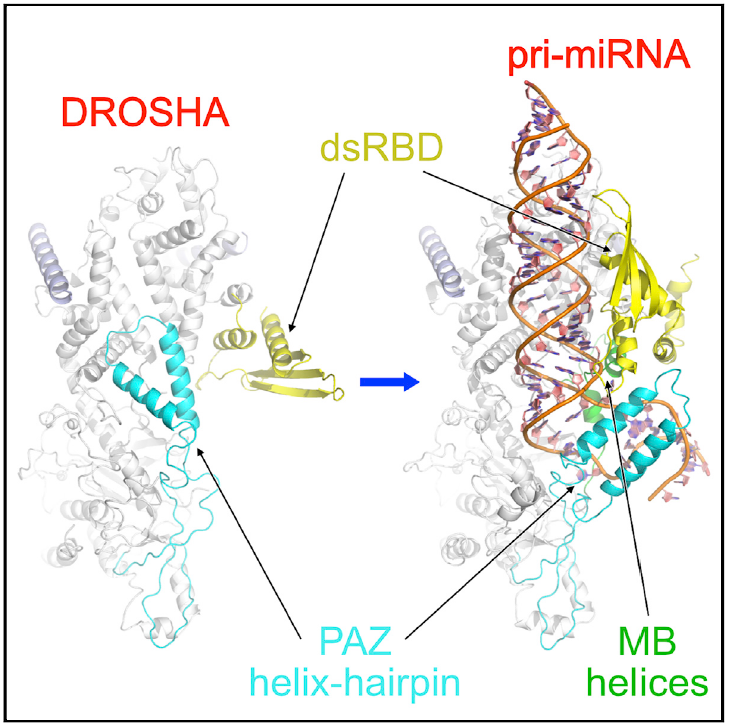
Drosha通过PAZ、MB helix、dsRBD结构域识别pri-miRNA,这些结构域在结合底物前(左)后(右)发生构象变化。
中国科学院生物物理研究所金文星博士,清华大学生命学院博士后王家为本文共同第一作者,清华大学生命学院王宏伟教授和中国科学院生物物理研究所所长许瑞明教授为本文共同通讯作者。本研究得到国家蛋白质科学研究(北京)设施清华大学基地、北京市结构生物学高精尖创新中心、北京市生物结构前沿研究中心的大力支持。
原文链接:
https://doi.org/10.1016/j.molcel.2020.02.024