清华大学生命学院鲁志课题组在《Nucleic Acids Research》杂志在线发表方法学论文题为《核糖体分析数据降噪处理及细胞翻译动态性特征的描述》(Ribosome elongating footprints denoised by wavelet transform comprehensively characterize dynamic cellular translation events)。该论文利用小波变换对核糖体分析(ribosome profiling, Ribo-seq)数据进行系统性降噪,开发了一套新型的鉴定RNA翻译活性的方法——RiboWave。
Ribo-seq是一种新型的高通量二代测序手段,通过特异性捕捉80S核糖体及其保护的RNA片段来检测全基因组水平蛋白翻译的状况。Ribo-seq的发明使得全基因组范围内的单碱基(single nucleotide resolution)的定量翻译成为可能。相比于传统研究蛋白翻译的手段,Ribo-seq还具有瞬时、定量和定位的优势。目前,Ribo-seq已经被广泛的应用于不同实验以及生理条件下蛋白翻译的研究中。与此同时,近年来也有越来越多的报道指出Ribo-seq数据存在大量的干扰信号。多重因素如测序误差、RNA片段污染、加药处理、RNA结合蛋白(RNA binding protein,RBP)结合以及P-site位点的定位不准确都会在Ribo-seq数据中引入不可避免的噪音。这些非翻译信号的掺杂将会在后续的分析过程中带来误差,使得研究工作人员无法准确地鉴定开放阅读框(open reading frame,ORF)的翻译活性,尤其是鉴定非编码RNA的蛋白编码潜能或特定条件下发生翻译的功能性短肽。此外,由于Ribo-seq信号无法代表真实的细胞翻译信号,直接通过原始数据来定位翻译起始位点变得较为困难,往往需要依赖额外的药物处理或其他手段实现。
针对此问题,鲁志课题组提出一个新的分析方法——RiboWave。RiboWave利用小波变换对Ribo-seq原始数据进行时域—频域的分解,提取原始信号中呈现3-nt周期性的翻译信号。它的优点在于可以对输入的原始信号进行多尺度细化分析,不仅可以从频率图谱中特异性提取3-nt周期,还可以推断出3-nt频率具体从哪个位点出现又从哪个位点消失,从而可以有效的区分翻译信号(3-nt)和非翻译信号(non 3-nt)。相比于原始信号,以降噪后的3-nt翻译信号为基础能够更加准确的描述ORF翻译过程,包括鉴定ORF的翻译活性以及定位翻译的起始位点。在与同类型其他方法的比较中,RiboWave表现出显著的优越性和稳定性。
不仅如此,RiboWave还被应用于研究细胞内动态的翻译调控行为,即降噪后的3-nt翻译信号可以参与细胞内蛋白质水平的定量、差异翻译行为的研究以及核糖体移码现象(ribosomeal frameshift)的鉴定等。总而言之,RiboWave方法是一个功能强大的Ribo-seq数据分析方法,通过提取细胞翻译特异性的3-nt周期性信号从而准确的还原真实的翻译活动并构建完整的细胞翻译谱图,帮助科研工作人员加深对细胞内翻译调控的认知和理解。该工具此前已向领域内同行公开,下载链接为https://lulab.github.io/Ribowave。在此基础上,实验室正在致力于结合RNA结构组数据,分别研究在逆境和肿瘤等重要生物学过程中RNA二级结构与蛋白翻译之间的协同关系。
鲁志实验室直博生徐知彧和胡龙为本文的共同第一作者,鲁志研究员为本文的通讯作者。该研究由国家重点研发计划、国家自然科学基金、北京市高精尖中心提供经费支持,数据的计算分析工作也得到了国家蛋白质科学研究(北京)设施清华基地生物计算平台的支持。
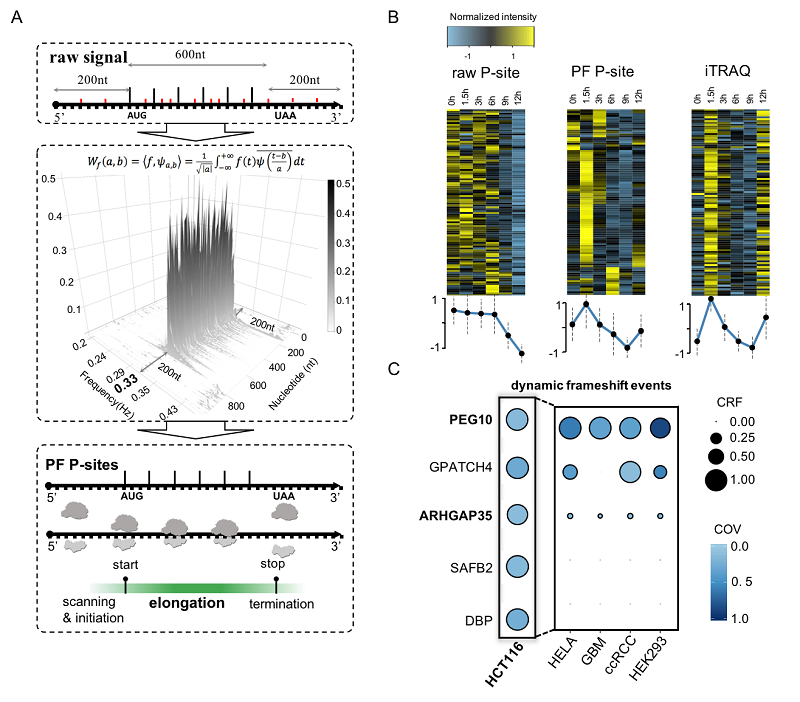
图1 RiboWave技术原理和应用实例
论文链接:https://www.ncbi.nlm.nih.gov/pubmed/29945224
软件链接:https://lulab.github.io/Ribowave