2019年3月19日,清华大学生命科学学院杨雪瑞课题组在Cell Reports发表大队列肿瘤多组学数据的深度整合分析工作:癌症特异性转录调控网络对启动子DNA甲基化组的依赖(Dependency of the cancer-specific transcriptional regulation circuitry on the promoter DNA methylome)。文章使用公开数据库癌症基因组图谱(The Cancer Genome Atlas,TCGA)的肿瘤多组学数据,采用基于信息论的数据挖掘策略,首次以CpG位点解析度系统阐述了21种主要癌症中启动子区DNA甲基化组在肿瘤基因转录调控网络中的深度参与。
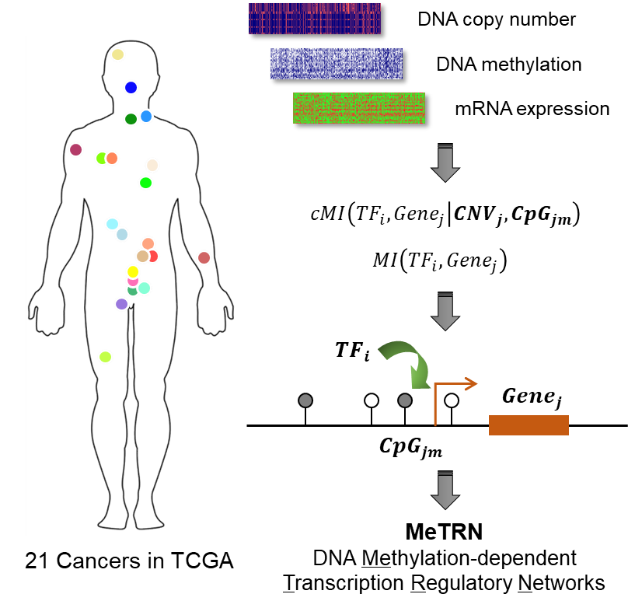
文章研究思路概念图。
转录因子对其靶基因的调控作用网络是基因表达调控的基本框架。但是,大量的研究表明转录调控并非简单的由转录因子到靶基因的简单二元调控模型。除转录因子外,转录过程中有多种“第三方”调控因子的协同参与,构成对基因转录过程的精密调控机制。已经发现一些长非编码RNA,DNA甲基化、DNA/RNA结合蛋白等均可能扮演该第三方调控因子的角色,但是这些协同调控因子尚未得到系统的鉴定,它们参与转录调控网络的具体机制也没有全面清晰的描述。
基因组及转录组学在过去十多年有了非常丰富的积累,特别是在癌症研究领域,多个国际协作项目对几乎所有主要肿瘤类型进行了非常广泛的多组学研究,产生了丰富的大数据资源。多组学生物大数据的深入挖掘及方法开发是杨雪瑞课题组的重要研究方向,作为成果之一,课题组开发了基于条件互信息概念的数据挖掘流程,用于系统鉴定一系列参与转录调控网络的第三方调控因子。
在此次发表的论文中,课题组聚焦各类肿瘤中高度特异的启动子DNA甲基化组,使用TCGA项目21种癌症中总计7000余例肿瘤组织的基因组、转录组、甲基化组等数据,全面评估了各DNA甲基化位点对转录因子和靶基因之间的调控关系的影响(见下图)。最终结果汇总为21种癌症中DNA甲基化参与的转录调控网络(Methylation-dependent Transcription Regulatory Network,MeTRN),并得到了来源于ENCODE项目的ChIP-seq数据的大规模验证。文章中的分析显示,DNA甲基化参与的转录调控是基因表达过程的核心调控层级之一,许多重要的癌症相关基因强烈依赖于这种调控机制。
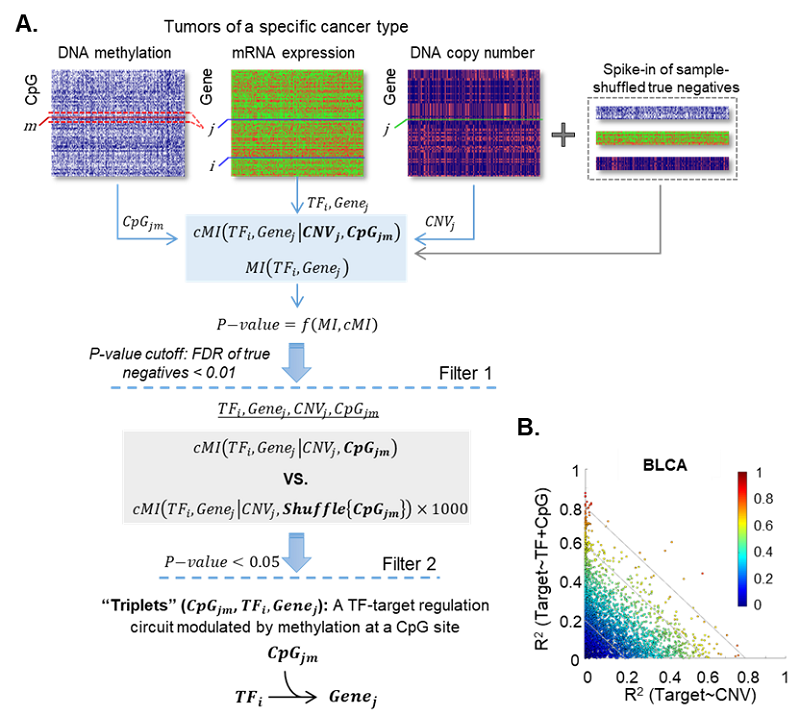
系统鉴定肿瘤中DNA甲基化参与的转录调控通路
A. 分析算法流程:使用肿瘤队列多组学数据鉴定DNA甲基化对转录调控通路的影响。B. 以膀胱癌(BLCA)为例,不同基因的表达水平可能有高度差异化的调控机制(甲基化参与的转录调控或拷贝数变异调控)。
基因组DNA的甲基化是细胞中最常见的表观遗传修饰之一,在细胞的分化及细胞身份维持中起重要作用,在各类癌症中也发生了大规模的异常,参与许多重要癌症相关基因的表达调控。本论文的工作是对DNA甲基化参与的转录调控过程的首次系统全面的描述,将转录因子与DNA甲基化组的耦合精确到单个CpG位点的尺度。文章的分析显示,甲基化位点及转录因子的耦合能够有效地对癌症病人的预后进行分类预测,提示了DNA甲基化位点及转录因子共同作为预后相关生物标志物的潜力。通过对DNA甲基化位点调控功能的全面普查,这项研究检验了DNA甲基化在不同癌症中参与转录调控网络作用的异同,为后续表观遗传组与转录组的整合研究提供了新的信息资源与理论支持。
本研究由CLS项目博士生刘昱、PTN项目博士生刘阳、硕士生黄荣耀共同完成,杨雪瑞为通讯作者。本研究由国家重点研发计划“精准医学研究”重点专项及国家自然科学基金委提供经费支持。清华大学蛋白质研究技术中心生物计算平台提供了项目所需大规模计算资源的支持。
文章链接:https://www.cell.com/cell-reports/fulltext/S2211-1247(19)30270-0
关于杨雪瑞:http://life.tsinghua.edu.cn/publish/smkx/11230/2018/20180205192912311488175/20180205192912311488175_.html